摘要
背景:复杂的染色体重排和染色体微缺失微重复综合征通常与患者异常的临床表型相关。2q31.1微缺失综合征十分罕见,可导致四肢及多重脏器的畸形。在本研究中我们揭示了造成一位疑似2q31.1微缺失综合征的4岁男孩身体及精神异常的遗传学基础。
方法:细胞遗传学及分子生物学技术,包括核型分析、微阵列比较基因组杂交技术(aCGH)、荧光原位杂交(FISH)和实时荧光定量PCR(real-time PCR),均被用于患者染色体异常的鉴定。
结果:患者染色体2q31.1区段存在一3.6Mb的缺失,同时2号、3号、6号、15号和18号染色体存在复杂的染色体重排。2q31.1区段染色体的缺失导致若干个已知致病基因拷贝数丢失,其中GAD1、DCAF17、SLC25A12和ITGA6的缺失与智力迟滞和面部畸形相关,DLX1/DLX2缺失部分造成四肢异常。另外两个基因HOXD13 和CHN1,是四肢和眼睛发育所必需的基因,在此例患者中虽具有正常的拷贝数,但是淋巴细胞的CHN1表达量却有所降低。
结论:我们推测2q31.1区段染色体的缺失和/或染色体易位对基因HOXD13 和CHN1的表达产生位置效应,造成这两个基因表达剂量不足。同时与2q31.1区段拷贝数缺失的致病基因相互作用,为患者复杂的临床表型提供了一个合理的解释。
1. 背景介绍
2q31.1微缺失综合征目前定义明确,且临床表型易于辨认[1,2]。患者临床表现多样,包含发育迟缓、智力迟滞、面部畸形、四肢残疾、心脏和眼睛缺陷、颅缝早闭、生殖器异常、癫痫和行为失调等[2-11]。本文报道了一例2q31.1区段存在3.6Mb缺失且另有5条染色体存在复杂染色体平衡易位的案例,遗传学分析和临床表型均表明该案例与2q31.1微缺失综合征相关,同时2q31.1下游区域存在一段来源于18号染色体的易位区段,共同导致此例患者复杂的临床表型。
2. 材料与方法
2.1 微阵列比较基因组杂交技术(aCGH)
采用Agilent公司的人类基因组CGH 244K 和I M两款芯片(Agilent公司,圣克拉拉,加利福尼亚州)进行患者染色体异常的鉴定。包含基因组DNA标记、杂交、洗涤和结果扫描在内的所有实验步骤均按照Agilent公司提供的标准流程进行。
2.2 荧光原位杂交(FISH)
采用FISH技术对细胞分裂中期染色体进行探针杂交,验证aCGH检测结果,所用探针为荧光标记的细菌人工染色体(bacterial artificial chromosome,BAC)探针(BlueGnome公司,剑桥,英国)。FISH探针靶向区域包括2号染色体的长臂区段(2q11.2,RP11-363D14,绿色)、18号染色体着丝粒区段(18p11.31,RP11-471M17,绿色)、特异染色体缺失区段(2q31.1,RP11-227L6,橙色)和特异染色体易位区段(CHN1特异)(2q31.1,RP11-493G24,橙色)。
2.3 反转录实时荧光定量PCR(RT-qPCR)
培养患者、患者母亲以及正常对照男性的淋巴细胞,提取细胞的mRNA用于基因表达量分析。反转录过程采用HiScript? 1st Strand cDNA Synthesis Kit(Vazyme公司,美国)进行。实时荧光定量PCR反应通过AceQ? qPCR SYBR Green Master Mix Kit(Vazyme公司,美国)来特异检测ITGA6、HOXD13 和CHN1的表达量。引物序列以及PCR条件详情参照试剂盒流程。
3. 结果
本研究中的4岁男性患儿,经亲子鉴定为一对非近亲结婚健康夫妇的唯一子女(数据未显示)。患儿母亲在怀孕的第一个月、第五个月和第六个月均有过感冒症状,但未发烧,也未采取药物治疗。孕38周超声检测未发现胎儿异常,但胎儿在孕38周时出现心跳过缓和子宫内窒息的症状,紧急剖腹产使其出生。患儿出生后14个月检测患儿的体重、身长、头围等参数均小于3SD(SD,标准方差),但发育商数(developmental quotient ,DQ)却从79降至36。
患儿面部畸形,眼睑狭小、眉间距宽、耳廓增厚。右足的第二、三趾存在并趾现象,左足底部存在一黄豆粒大小肉赘,左手大拇指外侧存在一多指。眼科检测发现患儿眼睛内斜视,下眼睑和结膜结合异常,泪道阻塞(图1)。患儿外生殖器发育正常,但阴茎短小。右手手掌有一横向的断掌纹,左手小指畸形。左右手无名指的关节和小指均出现屈肌和痉挛。四肢皮纹普遍均匀减少,双边膝盖呈平直状,X射线透视显示患儿仅有一个左腕骨,存在不间断的径向侧骨骺,尺骨临时钙化且半径增厚,骨和左拇指末端的纵向间隙轻度脱钙。
患儿45个月大时内分泌检测结果正常。但是,胰岛素生长因子(insulin-like growth factor-1,IGF-1)含量<25 ng/mL,低于正常水平(正常参考范围:49.00-289.00 ng/ml)。

图1. 患儿的临床特征图。眼内斜视、眼距宽(A),眉眼间距宽(B),耳廓增厚(C),躯干和四肢的X-射线透视图(D,E),手指末端关节浮动(F),并趾(G),X-射线透视下的足异常(H)
常规核型分析显示患儿父母染色体正常(数据未显示)。高分辨率的核型分析显示患儿染色体核型为46,XY,t(2;3;6;15;18),del(2)(q),2号、3号、6号、15号和18号染色体存在复杂的染色体重排:46,XY,del(2)(q31 → q32),t(2;3;15)(2qter →2q35::3q27 → 3q21::2p13 → 2q31::15q15 → 15qter;15pter →15q15::2p13 → 2pter),t(2;3;6;18)(3pter → 3q21::18q21.1 →18qter;3qter → 3q27::6p23 → 6qter; 18pter → 18q21.1::2q31→2q35:: 6p23 → 6pter)(图.2A,B,C)。染色体的断裂和重接如下:6号染色体的一段易位至18号染色体,18号染色体的一段易位至3号染色体,3号染色体的一段易位至2号染色体。2号染色体发生三处断裂,非着丝粒的三处断裂区段分别易位至6号、15号和18号染色体。2号染色体剩余的着丝粒区段与来自于3号、15号染色体的区段重新组成新的2号染色体。值得一提的是,我们在2q31.1区段发现一处断裂,其上游的2q31区段与15q15 → 15qter重新组合,下游的2q31 → 2q35区段与18q21.1 → 18pter重新组合。
为了确认是否存在由于染色体重排导致的染色体不平衡,我们利用Aglient CGH 244K芯片对患者DNA进行检测,确认患者在2q31.1 (171,383,345–174,989,964)区段存在缺失,该缺失进一步通过Aglient CGH 1 M芯片在chr2:171383345–175015341位置得到确认(图.2D)。2q31.1区段缺失大小3.6Mb,包含21个基因,其中GAD1、DCAF17、SLC25A12、ITGA6与疾病相关(http://www.ncbi.nlm.nih.gov/omim/)。2q31.1区段缺失通过FISH检测得到了验证(图.2E)。2号染色体对照探针RP11-363D14 (2q11.2)与正常2号染色体和衍生2号染色体均有杂交,但是2q31.1区段探针RP11-227L6仅与正常2号染色体杂交,不与衍生2号染色体杂交。探针RP11-493G24与正常2号和衍生2号染色体均杂交,表明2q31.1区段的CHN1基因不存在拷贝数变化(图.2F)。
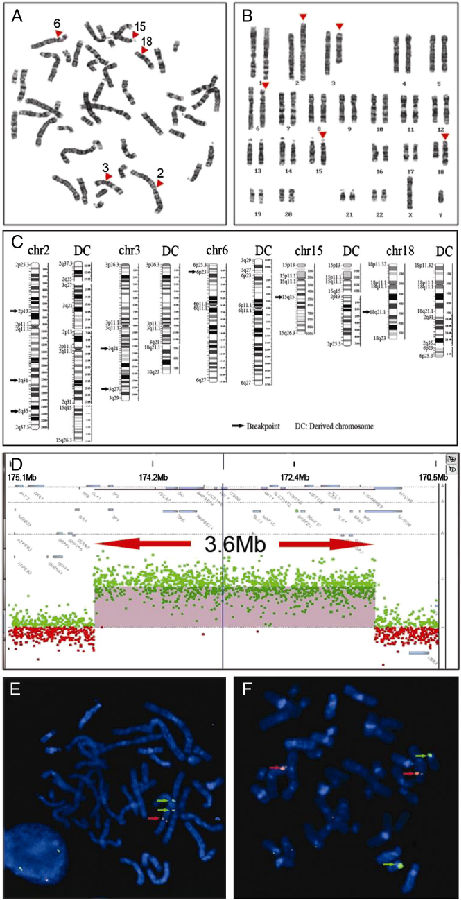
图2. 利用细胞遗传学和分子生物学分析患儿的DNA。核型分析G显带技术(A),核型分析的象形图(B),染色体结构重组模型(C),Agilent 1 M aCGH结果图(D),双重FISH结果图(E和F)。涉及2号、3号、6号、15号和18号染色体的多重染色体重排用箭头显示。D图的红色箭头显示2q31.1区段缺失开始和结束的放大图。E图的红色箭头显示探针BAC RP11-227L6 (2q31.1)的杂交信号,仅有一个杂交信号,表明存在2q31.1缺失;绿色箭头表明探针RP11-363D14 (2q11.2)的杂交信号。F图中红色箭头显示探针BAC RP11-493G24(含基因CHN1,2q31.1)的杂交信号,两个杂交信号表明基因CHN1未缺失;绿色箭头显示探针RP11-471M17 (18p11.31)的杂交信号。(对于此图中颜色的注释,读者可以参考此文的网络版)
对于RNA表达量的分析显示,基因ITGA6、CHN1和HOXD13在患者淋巴细胞的表达量显著低于其母亲和作为对照的正常男性(图3),基因HOXD13的表达量甚至低至无法精确测定。
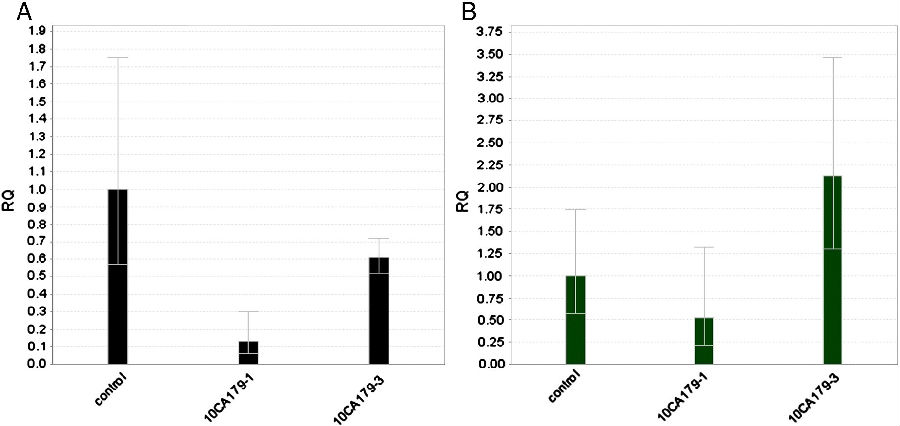
图3. 实时荧光定量PCR分析ITGA6(A)和CHN1(B)在淋巴细胞的表达量。10CA179-1(患者),10CA179-3(患者母亲)和对照(正常男性)
4. 讨论
本研究探讨了一位4岁男孩身体及精神异常的遗传学基础。这也是文献报道的首例2q31.1微缺失且伴随2号、3号、6号、15号和18号五条染色体重排的案例。通过核型分析、aCGH和FISH的方法,我们发现了新发的同源染色体间复杂罕见的染色体重排46,XY, t(2;3;6;15;18),同时伴随2q31.1区段的染色体微缺失。有意思的是,患儿的父母双亲染色体正常,因而我们推测在受精卵着床前,合子未激活时发生了这种复杂的染色体重排,随后进行产前发育。具体染色体断裂和重接的原因不明,不过推测与复制过程中DNA修复系统失效、接触致畸的环境或药物以及蛋白质/组蛋白的结构扰动相关[12]。包括本案例在内的大多数复杂染色体重排案例都伴随一系列的临床表型,包括智力低下和多重先天畸形等[13,14]。
本研究强调了利用多项分子生物学技术,如FISH、aCGH和qPCR等研究染色体异常的分子机制,并将基因型与表型建立关联的重要性。aCGH的检测结果发现,除2q31.1存在缺失外,复杂的染色体重排并未造成染色体的不平衡。因而我们推测2q31.1区段的缺失是造成患儿临床表型的主要因素。以往的研究已经报道了一系列与2q31.1缺失相关的临床表型,如发育迟缓、智力迟滞、面部畸形、先天性心脏病、四肢畸形、生殖器官异常、颅缝早闭以及癫痫等[1]。分子生物学分析发现2q31.1区段缺失造成21个基因的丢失,其中15个基因的丢失造成一名19岁女孩患有严重的渐进型自闭症[15],本研究中的4岁男孩另外还丢失了基因MYO3B、SP5、GAD1、GORASP2、TLK1和METTL8 ,这两名患者均表现为智力迟滞。在两名患者均丢失的15个基因中,DCAF17、SLC25A12 和ITGA6与疾病相关,其中DCAF17 (OMIM 241080)与Woodhouse–Sakati综合征相关,表现为性腺机能减退、脱发、糖尿病、精神发育迟滞和锥体外系综合症等[12],SLC25A12 (OMIM 612949)则与全脑髓鞘化低下相关[16],而ITGA6 (OMIM 226730)则与幽门闭锁关联的结大疱性表皮松懈相关[17]。患病男孩表现出的面部畸形、生长和智力发育迟滞便由上述基因拷贝数缺失引起。
本研究中的患病男孩还存在典型的骨骼畸形、多指畸形和并趾。有报道位于小鼠染色体2q31.1的DLX1/DLX2(远端缺失)对颅面、四肢和骨骼的发育十分重要[15]。虽然之前报道的19岁患病女孩DLX1/DLX2也存在拷贝数缺失,但未发生骨骼畸形[15],我们推测患病男孩呈现的其他畸形是2q31.1区段缺失和染色体易位共同作用的结果,它们也协同引起患儿骨骼和四肢的畸形。此外,患病男孩还存在眼斜视、眼睑低下和泪管畸形等眼睛畸形,无法用2q31.1区域内任何基因缺失进行解释。
为了找寻用于解释患病男孩临床症状的其他候选基因,我们挑选了距离2q31.1缺失区段近端和远端的两个基因HOXD13 和 CHN1进行研究。HOXD13位于2q31.1区段的远端,已有报道其与先天的并指/趾、多指/趾征相关[6],而CHN1则在大脑和眼睛的发育,以及神经信号的传导和应答中起重要作用[18,19]。aCGH检测结果显示包含基因HOXD13 和CHN1的区段未发生染色体拷贝数的变化,这一结果也通过FISH得到了验证。既然基因HOXD13和CHN1不存在缺失,我们推测2q31.1区段的缺失和/或2q31.1–q35区段对18号染色体易位产生的位置效应,导致了基因HOXD13和CHN1表达量的下降。患儿淋巴细胞中CHN1表达量的下降也支持这一推测。众所周知,染色体的易位和倒位可通过引入新的启动子、增强子或抑制子而影响邻近基因的表达[20],因而我们进一步推测,本案例中染色体的易位导致了基因HOXD13和CHN1表达量下降,其中,HOXD13表达量下降导致患病男孩骨骼和四肢畸形,CHN1表达量下降导致患病男孩眼睛畸形。若进一步验证此推测,需要利用第二代测序技术找寻染色体易位的断裂点,同时进行相关组织切片基因表达量的检测。
综上,本研究报道了一例2q31.1微缺失综合征同时伴随复杂的染色体平衡易位的案例,证实了多重分子生物学技术,如FISH、aCGH、qPCR在研究复杂临床表型和具体微缺失综合征之间的关联时具有积极作用。本案例的染色体重排可通过几种机制产生,如非等位基因同源重组(NAHR)[21]和非同源末端连接(NHEJ)[22]。另外,复制叉迟滞和模板转换(FoSTeS)也可能产生另一种基于基因复制的染色体重排机制[23]。不过,本研究仅是针对患者的染色体异常和临床表型进行研究,若要进一步了解此患者染色体异常产生的原因,则需要进行更为详细的检测,如采用第二代测序技术确认染色体缺失和易位的位点,以及对所有相关疾病的组织切片进行转录水平的分析。
作者公开声明
致谢
参考文献
[1] Dimitrov B, Balikova I, de Ravel T, et al. 2q31.1 microdeletion syndrome: redefining the associated clinical phenotype. J Med Genet 2011;48:98–104.
[2] Maas SM, Hoovers JM, van Seggelen ME, et al. Interstitial deletion of the long arm of chromosome 2: a clinically recognizablemicrodeletion syndrome? Clin Dysmorphol 2000;9:47–53.
[3] Boles RG, Pober BR, Gibson LH, et al. Deletion of chromosome 2q24–q31 causes characteristic digital anomalies: case report and review. Am J Med Genet 1995;55:155–60.
[4] Nixon J, Oldridge M, Wilkie AO, et al. Interstitial deletion of 2q associated with craniosynostosis, ocular coloboma, and limb abnormalities: cytogenetic andmolecular investigation. Am J Med Genet 1997;70:324–7. 284
[5] Slavotinek A, Schwarz C, Getty JF, et al. Two cases with interstitial deletions of chromosome 2 and sex reversal in one. Am J Med Genet 1999;86:75–81.
[6] Del Campo M, Jones MC, Veraksa AN, et al. Monodactylous limbs and abnormal genitalia are associated with hemizygosity for the human 2q31 region that includes the HOXD cluster. Am J Hum Genet 1999;65:104–10.
[7] Carranza Rojo D, Hamiwka L, McMahon JM, et al. De novo SCN1A mutations in migrating partial seizures of infancy. Neurology 2011;77:380–3.
[8] Pereira S, Vieira JP, Barroca F, et al. Severe epilepsy, retardation, and dysmorphic features with a 2q deletion including SCN1A and SCN2A. Neurology 2004;63:191–2.
[9] Bijlsma EK, Knegt AC, Bilardo CM, et al. Increased nuchal translucency and split- hand/foot malformation in a fetus with an interstitial deletion of chromosome 2q that removes the SHFM5 locus. Prenat Diagn 2005;25:39–44.
[10] Langer S, Geigl JB, Wagenstaller J, et al. Delineation of a 2q deletion in a girl with dysmorphic features and epilepsy. Am J Med Genet A 2006;140:764–8.
[11] MencarelliMA, Caselli R, Pescucci C, et al. Clinical andmolecular characterization of a patient with a 2q31.2–32.3 deletion identified by array-CGH. Am J Med Genet A 2007;143A:858–65.
[12] Roukos V, Burman B, Misteli T. The cellular etiology of chromosome translocations. Curr Opin Cell Biol 2013;25:357–64.
[13] Batanian JR, Eswara MS. De novo apparently balanced complex chromosome rearrangement (CCR) involving chromosomes 4, 18, and 21 in a girl with mental retardation: report and review. Am J Med Genet 1998;78:44–51.
[14] Lee NC, Chen M, Ma GC, et al. Complex rearrangements between chromosomes 6, 10, and 11 with multiple deletions at breakpoints. Am J Med Genet A 2010;152A:2327–34.
[15] de Bruijn DR, van Dijk AH, Pfundt R, et al. Severe progressive autism associated with two de novo changes: a 2.6-Mb 2q31.1 deletion and a balanced t(14;21)(q21.1; p11.2) translocation with long-range epigenetic silencing of LRFN5 expression. Mol Syndromol 2010;1:46–57.
[16] Wibom R, Lasorsa FM, Tohonen V, et al. AGC1 deficiency associated with global cerebral hypomyelination. N Engl J Med 2009;361:489–95.
[17] Ashton GH, Sorelli P, Mellerio JE, et al. Alpha 6 beta 4 integrin abnormalities in junctional epidermolysis bullosawith pyloric atresia. Br J Dermatol 2001;144:408–14.
[18] Buttery P, Beg AA, Chih B, et al. The diacylglycerol-binding protein alpha1-chimaerin regulates dendritic morphology. Proc Natl Acad Sci U S A 2006;103:1924–9.
[19] Miyake N, Demer JL, Shaaban S, et al. Expansion of the CHN1 strabismus phenotype. Invest Ophthalmol Vis Sci 2011;52:6321–8.
[20] Kleinjan DA, van Heyningen V. Long-range control of gene expression: emerging mechanisms and disruption in disease. Am J Hum Genet 2005;76:8–32.
[21] Emanuel BS, Shaikh TH. Segmental duplications: an ‘expanding’ role in genomic instability and disease. Nat Rev Genet 2001;2:791–800.
[22] LieberMR. Themechanismof double-strandDNA break repair by the nonhomologous DNA end-joining pathway. Annu Rev Biochem 2010;79:181–211.
[23] Zhang F, Carvalho CM, Lupski JR. Complex human chromosomal and genomic rearrangements. Trends Genet 2009;25:298–307.
 妇产科在线APP下载
妇产科在线APP下载













