编者按

文献原文
拷贝数变异测序技术检测人类植入前胚胎染色体非平衡易位的验证研究
Li Wang,5,6David S. Cram,3,5,7 Jiandong Shen,5,8 Xiaohong Wang,5,9Jianguang Zhang,7 Zhuo Song,7 Genming Xu,7 Na Li,9Junmei Fan,6 Shufang Wang,10 Yaning Luo,9 Jun Wang,9Li Yu,10 Jiayin Liu,4,8 and Yuanqing Yao2,6
6 中国人民解放军总医院妇产科,中国北京;
7 北京贝瑞和康生物技术有限公司,中国北京;
8 南京医科大学第一附属医院生殖医学中心,生殖医学国家重点实验室,中国南京;
9 第四军医大学唐都医院生殖中心,中国西安;
10 中国人民解放军总医院血液科,中国北京
摘要
通过辅助生殖技术(assisted reproductive technologies,ART)所获得的胚胎常发生染色体非整倍体异常,这是导致胚胎植入失败及流产的重要原因。目前,胚胎植入前遗传学诊断(Preimplantation genetic diagnosis,PGD)通过微阵列技术确定可供移植的整倍体胚胎。我们设想将二代测序技术与复杂生物信息学算法联合应用,有望提供更加全面准确的方法,以提高胚胎检测的总体效率。为实现这一目的,我们开发出一种适用于单细胞分析的高分辨率拷贝数变异测序方法(Copy number variation sequencing,CNV-Seq)。在本验证研究中,用于CNV-Seq 检测的是24个PGD胚胎的全基因组扩增样本,这些样本经由金标准微阵列比较基因组杂交技术(array comparative genomic hybridization,Array CGH)确诊。结果显示,CNV-Seq 在检测染色体整倍体、非整倍体以及非平衡性易位方面具有高度的灵敏性和特异性。此外,CNV-seq也可用于易位染色体异常分离引起的小至1Mb的末端非平衡易位检测、比对以及精确定量。这些验证研究显示,CNV-Seq 进行胚胎检测具有准确可靠的特点,有望进一步提高 PGD 的总体效率。
关键词:
Array CGH,卵裂球活检,测序方法检测拷贝数变异,胚胎,胚胎植入前遗传学诊断
前言
1992 年,研究人员首次报道利用 PCR 法分析单卵裂球样品的男性特异SYR 基因,可用于 X 染色体连锁疾病的胚胎植入前遗传学诊断(Preimplantation genetic diagnosis,PGD)[1]。此后,诸多分子生物学技术用于染色体异常检测[2]。单细胞的荧光原位杂交(fluorescent in situ hybridization,FISH)最初被用于胚胎植入前遗传学筛查(preimplantation genetic screening,PGS),以检测与自发流产相关的染色体非整倍体(13号、16号、18号、21号和22 号以及 X 和 Y)[3]。针对染色体易位,设计特异性着丝粒与端粒探针,区分染色体易位携带者产生的平衡与非平衡易位胚胎,可减少自发性流产的发生[4]。FISH在同时检测24 条染色体方面存在不足,而全基因组扩增技术(Whole genome amplification,WGA)则可弥补这些不足,且可与寡核甘酸PCR[5] 、比较基因组杂交技术(Comparative genomic hybridization,CGH)联合运用。近年来,其它替代技术陆续出现,并被广泛用于胚胎植入前全部染色体非整倍体以及结构异常的检测,如微阵列比较基因组杂交技术(array comparative genomic hybridization,Array CGH)[7]、单核苷酸多态性微阵列技术(Single nucleotide polymorphism array,SNP array)[8,9] 和多重荧光定量 PCR[10]等。
FISH技术对PGD 周期中的配子及胚胎[3,11]进行检测,发现染色体非整倍体是由第一次减数分裂、第二次减数分裂以及受精后有丝分裂发生错误而引起,可导致早孕期胎停育、植入失败或自发性流产等。近期,微阵列技术对卵裂期胚胎进行分子生物学分析[12,13],由于人类胚胎染色体内在的不稳定性[14],其它类型的染色体异常也可发生,如染色体片段重复或缺失以及一些更复杂的结构异常。在临床实践中,Array CGH 与 SNP array 在检测染色体异常方面也显示出较高可靠性和准确性。然而,目前市场上的多种微阵列芯片平台在探针设计、探针定制以及探针密度分布方面存在显著差异。同时,由于可能存在等位基因随机丢失现象,某些平台在检测胚胎具有临床意义的染色体断裂方面,分辨率不高。此外,SNP array 对于检测染色体三体以及其它无减数分裂重组的染色体重复也存在不足[2]。而且,从样品活检到出具报告,微阵列芯片技术工作流程繁琐,需要进行 DNA 标记及杂交。除此之外,微阵列芯片及试剂价格高昂,且芯片探针数量有限,难以进行高效的检测。第二代测序技术(Next-generation sequencing,NGS)结合生物信息学算法,对IVF胚胎进行染色体异常检测,更加全面且经济有效,为 PGD 检测带来全新机遇。
以往研究证实,NGS 对于极体[15]、卵裂球[16,17] 以及囊胚滋养外胚层细胞[18] 的染色体检测具有可行性。但上述研究并未论述低覆盖测序深度下NGS的检测分辨率,以及是否可对拷贝数进行定量等问题,而这些对于胚胎活检样品的染色体分析至关重要。本研究小组研发出一种第二代测序检测拷贝数变异(Copy number variation sequencing,CNV-Seq)的新方法以解决这一问题。CNV-Seq的建立基础是全基因组测序技术及生物信息学分析方法在胎儿染色体非整倍体的无创性产前检测(NIPT)中得到验证[19]。经后续研究证实,CNV-Seq可检测最低嵌合比例为5%的母体染色体嵌合体[20],也可用于限制性胎盘嵌合的嵌合程度和性质分析[21,22],以及评估单细胞样品的全部染色体非整倍体状况[16]。
为检测胚胎活检样本,本研究对CNV-Seq 进行针对性改良,将基于PCR过程的WGA引入现有测序流程,以获得足量的 DNA 模板用于检测,同时通过复杂的数据分析,确定每条染色体上的平均CNV值。利用回顾性、双盲实验设计,评估CNV-Seq 对于胚胎单卵裂球样品WGA 产物的检测效果。所有样品均经Array CGH 确认其染色体异常情况。结果显示,CNV-Seq 和 Array CGH 具有高度的检测一致性。
材料与方法
实验设计
本研究PGD检测在三家单位进行:中国人民解放军总医院妇产科(北京),相关研究获得中国人民解放军总医院伦理委员会的批准(S2013-092-02);西安第四军医大学唐都医院生殖中心(西安),相关研究获得唐都医院伦理委员会的批准;南京医科大学生殖医学国家重点实验室(南京),相关研究符合 PGD 许可证要求。所有患者在接受Array CGH或CNV-Seq进行PGD检测时,均需签署知情同意书。用于CNV-seq检测的样本经由BlueGnome array CGH 确认其染色体状况。本研究CNV-seq对10 例样品进行回顾性检测,14例样品进行双盲检测。
IVF、胚胎活检和胚胎冷冻保存
VF及胚胎活检等操作均在中国人民解放军总医院妇产科及第四军医大学唐都医院进行,具体步骤参见文献[23]。采用卵细胞浆内单精子注射术(intracytoplasmic sperm injection,ICSI)对细胞分裂中期的卵细胞进行受精,避免多余精子造成污染。受精完成后(第 0 天),将受精卵置于 Vitrolife 公司生产的 G1 培养基中培养,15~18 小时后检测是否产生两个原核,以此判断是否受精。用磷酸盐缓冲液(Phosphate-buffered saline,PBS)洗涤受精卵,然后置于四孔培养板中(每孔一枚受精卵),滴加一滴 G1 培养基并用矿物油覆盖,于 5% CO2 培养箱中培养。受精卵培养 48 小时后进入卵裂期,更换 G2 培养基后继续培养,48~72 小时后进入囊胚期。卵裂期活检时,将卵裂球置于 20 μl Vitrolife 公司生产 G-MOPS 培养基中,并用矿物油覆盖。借助 Saturn 公司生产的非接触式激光仪打孔,显微操作下,使用拉细的玻璃吸管从每个卵裂球中获取一个卵裂球细胞。囊胚期活检时,使用摩擦分离法获取透明带上由于激光打孔(第 3 天)而外凸的 5~8 个滋养外胚层细胞。使用 PBS 洗涤细胞,并转移至含 2-4 μl 无菌 PBS 的 PCR 试剂管中。南京医科大学生殖医学中心运用类似方法进行 IVF 和胚胎活检[24]。
利用玻化技术将活检后的囊胚期胚胎冷冻保存。胚胎冷冻及解冻所用溶液均按生产商 Kitazato Supply 提供的说明步骤配置。首先将胚胎置于平衡液中,室温平衡 5 分钟,然后转移至玻化液中孵育 5 分钟,随后将胚胎放入 Kitazato Supply 公司生产的 Cryotop MVC 板中并立即置于液氮中。如冷冻胚胎经 PGD 诊断为整倍体,则将其 MVC 板浸泡入解冻液中,于 37℃ 放置 1 分钟解冻。将复苏胚胎转移至稀释液中放置 3 分钟,然后于洗涤液中孵育 10 分钟。经过 2 小时培养,将 1~2 枚胚胎放入胚胎导管中,植入母亲子宫内。
植入前遗传学诊断(PGD)
胚胎活检后用 PBS 洗涤三次单卵裂球细胞或滋养外胚层细胞。借助显微操作技术,将细胞小心转移至含有2.5 μl PBS的200 μl PCR管中。于 -20℃ 冷冻细胞备用。使用 PicoPLEX Kit 对活检后卵裂球细胞或滋养外胚层细胞以及 PBS 空白对照进行 WGA 扩增。从50 μl WGA 反应体系中取出 5 μl 扩增产物进行 1.5% 溴化乙锭琼脂糖凝胶电泳分析,验证WGA产物的质量及片段大小分布。用于文库构建的WGA 产物需满足两个条件,首先平均片段大小为300bp(100~500 bp),其次单卵裂球细胞WGA 产物浓度大于 50 ng/μl,滋养外胚层细胞WGA 产物浓度大于 100 ng/μl。分别用 Cy5 和 Cy3 对对照及待测 DNA 样品进行标记,然后根据 Array CGH 标准操作步骤,用 BlueGnome 公司生产的 24sure V2 或 V3微阵列芯片检测胚胎染色体状况[7]。
CNV-seq及数据分析
利用 Zymo Research 公司的 DNA Clean & Concentrator Columns 纯化 WGA 产物,去除未结合的引物后重悬制成 50 ng/μl 溶液。按文献方法[19,25],构建末端含有测序引物及9 bp 序列标签的测序文库,然后用 Illumina HiSeq 2500平台测序,产生约 500 万条36 bp 单端测序序列,基因组覆盖度0.06倍。用 Burrows-Wheeler 算法分析测序结果,将其比对到未去重复的参考基因组上[26]。将约 300 万条唯一匹配的测序序列分配到 2万个连续的测序单元中(每个测序单元约30条测序序列)。按文献方法,对同一测序过程中至少 15 个测试样品进行内部比对[20]。用 CNV-seq 拷贝数算法[27,28]估算每条染色体上平均 CNV值。
以每2万个测序单元的标准化测序密度的 log2 值为y 轴,以连续 2 万个测序单元为X轴作图,得到 24 条染色体的 CNV-Seq 检测结果。图中曲线上红色方框表示重复序列区域,黑色方框表示着丝粒,上虚线 [log2 (3/2)]表示100%染色体增加(重复),下虚线 [log2 (1/2)]表示100%染色体减少(缺失),蓝线表示每条染色体上平均的CNV值,可相对 hg19 参考基因组定量及映射各区域的CNV。
结果
CNV-Seq 检测染色体非整倍体的有效性
Array CGH 是目前PGD检测胚胎染色体异常的金标准[7]。本研究以BlueGnome BAC Array CGH检测结果作为参考,评估CNV-seq对于胚胎染色体异常的检测效能。共有10份WGA产物用于CNV-seq的有效性研究,其中8份来自于非整倍体胚胎样本,另有2份来自于已移植且生出健康足月儿的整倍体胚胎样本。结果显示,CNV-Seq取得与 Array CGH 完全一致的检测结果(表1)。本文列举了2例Array CGH 与 CNV-Seq 检测结果一致的样本,分别为2 号染色体单体胚胎和22 号染色体三体及 7q 部分单体胚胎(图1;附录图S1为24 条染色体的检测结果;数据信息可在www.biolreprod.org查找)。综上,回顾性研究显示Array CGH 与 CNV-Seq对染色体非整倍体的检测一致性为 100%(10/10)。
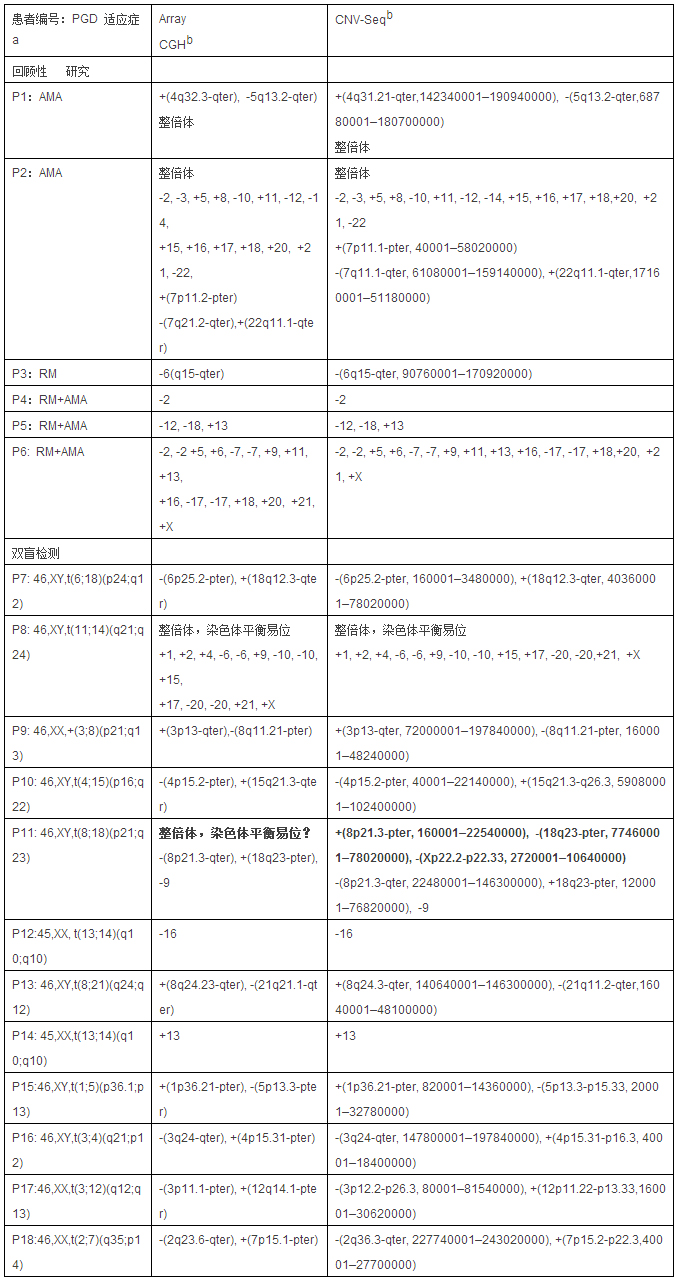
aRM,反复流产;AMA:高龄孕妇 b染色体缺失与重复分别用 - 和+ 表示。括号显示CNV-Seq检出的染色体非平衡易位片段在hg19 参比基因组上的对应坐标。粗体表示 Array CGH 与 CNV-Seq 检测不一致。
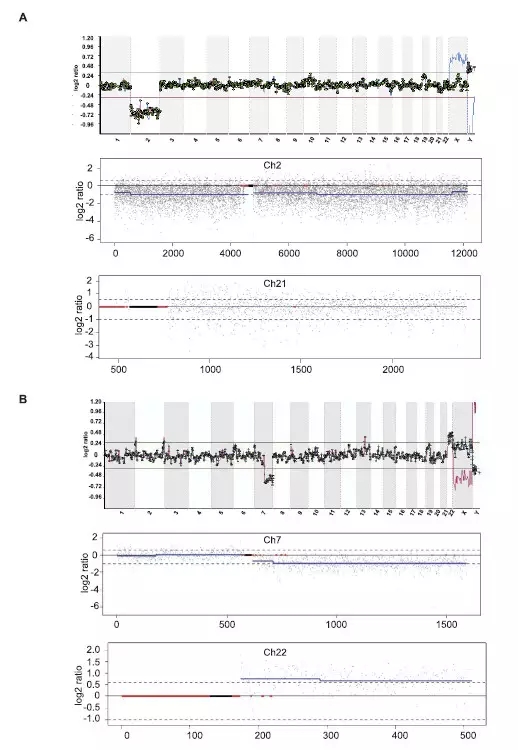
图1 Array CGH 与 CNV-Seq 对染色体非整倍体胚胎的检测一致性。A) 2 号染色体单体,以正常21号染色体作为对照。B)22号染色体三体及7q部分单体。
CNV-Seq 检测染色体非平衡易位的有效性
CNV-seq针对染色体非平衡易位的有效性研究在南京医科大学第一附属生殖医院临床中心进行,仍使用 BlueGnome BAC array CGH 作为评估CNV-seq检测结果的参照。共有来自于12个不同案例的14份WGA产物用于CNV-seq检测,后续分析CNV-seq与Array CGH对比结果(表1)。14份样品中,CNV-Seq 正确检出经Array CGH确认过的9 例染色体非平衡易位,另检出1例9 号染色体单体。本文列举了染色体t(3;8)(p21;q13) 相互易位患者的CNV-Seq检测结果,发现预期断裂点存在两个非平衡延伸,包括3p13-qter区段126 Mb 的重复和 8q11.21-pter区段32.2 Mb 的缺失(图2;附录图S2为24条染色体的检测结果)。
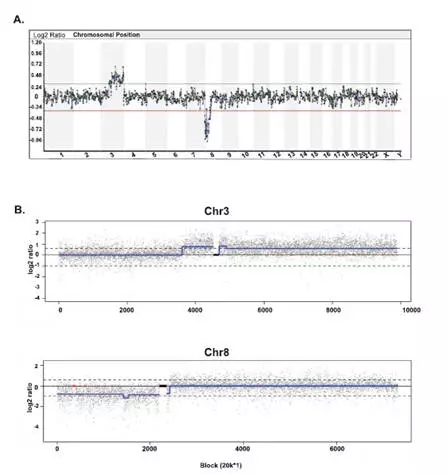
图2 Array CGH 与 CNV-Seq 对染色体非平衡易位胚胎的检测一致性。此案例3 号与 8 号染色体因第一次减数分裂期间发生相邻分离而导致t(3;8)(p21;q13) 非平衡易位。Array CGH 与 CNV-Seq 均显示p13-qte区段存在 126 Mb 重复,8q11.21-pter区段存在 32.2 Mb 缺失。
不过Array CGH 和 CNV-Seq在对t(8;18)(q21;q23) 相互易位且18 号染色体断裂点靠近q 端的胚胎检测时,检测结果不一致。Array CGH检测该胚胎正常(图3),但CNV-Seq 发现该样品存在两处非平衡易位,并明确检出8p-qter区段34 Mb 的重复、18-qter区段0.9 Mb的缺失和 Xp 末端7.9 Mb 的缺失。CNV-Seq正确检出其余4枚胚胎的非平衡罗伯逊易位、单纯性和复合性染色体非整倍体。重要的是,本研究唯一纳入的一枚整倍体胚胎也被CNV-Seq 正确检出(表1)。综上所述,Array CGH 和 CNV-Seq对染色体非平衡易位的检测一致性为93%(13/14)。其中,Array CGH 漏诊一例染色体非平衡易位,且在一例与 CNV-Seq检测结果相符的案例中漏诊Xp末端7.9 Mb 的缺失。
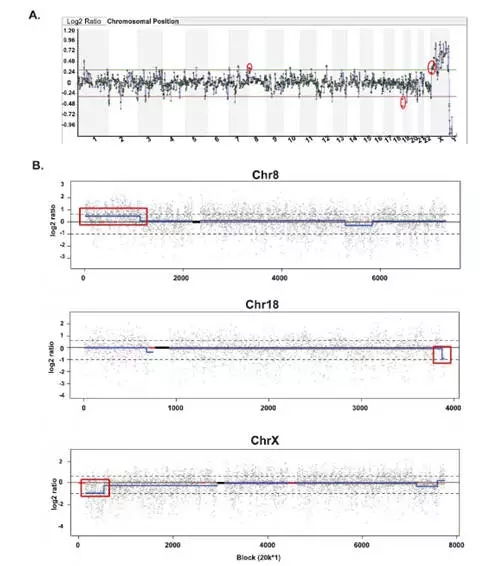
图3 CNV-Seq 对 Array CGH 漏检的染色体t(8;18)(p21;q23) 非平衡易位胚胎的检测。Array CGH 检测该胚胎正常,但怀疑存在染色体非平衡易位(红色圆圈表示检测探针)。CNV-Seq 则明确检出因第一次减数分裂期间发生 t(8;18)(p21;q23) 易位而导致的8p-qter区段34 Mb 的重复、18-qter区段0.9 Mb 的缺失,以及Xp末端7.9 Mb 的缺失。
讨论
本研究验证CNV-seq对IVF 胚胎染色体异常进行 PGD检测的有效性。通过回顾性临床试验和双盲检测,直接比较 Array CGH 与 CNV-Seq 对相同单卵裂球 WGA 产物的检测效果,以验证 CNV-Seq 的检测效能。CNV-Seq 成功检出了3 枚整倍体胚胎、12 枚全部或部分非整倍体胚胎,以及9枚不同片段非平衡易位胚胎(因亲代染色体错误分离导致)。1枚胚胎的Array CGH 和 CNV-Seq 检测结果不一致,Array CGH 检测为正常,而CNV-Seq 则发现8号 和 18号染色体存在非平衡易位。此外,CNV-Seq 还正确判断出所有 25 枚胚胎的性别。有效性研究结果显示,CNV-Seq 可用于检测全部或部分染色体非整倍体,以及由染色体错误分离造成的末端非平衡易位,其检测能力与 BlueGnome array CGH 相当。当然,在 CNV-Seq 被纳入常规 PGD检测之前,还需进行大量胚胎样本的更多临床研究,以确认CNV-Seq的检测灵敏度和特异度。
在以往研究中,我们运用低覆盖度高通量测序方法分析待测样本的染色体异常,从而实现了全染色体非整倍体的准确判断[16]。本研究引入更复杂的生物信息学算法,将测序片段从较小的染色体片段中区分出来并逐步分析,从而提高了检测分辨率,可实现对CNV的识别。为了将这一高分辨率引入 CNV-Seq并进行胚胎分析,将低覆盖度测序技术随机产生的约300 万条测序片段,沿每条染色体长度映射到20 kb的连续测序单元中(每个测序单元约 30 条测序片段),从而在数据分析时最大限度地使用每一条测序片段提供的信息。本质上,CNV-Seq 使用约 150000 个数据点对 24 条染色体进行分析,是BlueGnome 微阵列芯片3000个探针的50 倍,后者提供的基因组平均分辨率仅为1 Mb[7]。通过比较,我们认为 CNV-Seq 对24 条染色体均可提供高分辨率的检测(含高度重复区域的 Y 染色体以及近端着丝粒染色体的 p 短臂除外)。此外,CNV-Seq 还可更准确地绘制出相对于 hg19 参比基因组的 CNV图谱 [26]。由于 CNV-Seq 在相同测序深度下具有较高分辨率,其临床应用可从全染色体非整倍体[16]进一步扩展至小片段染色体非平衡易位及 CNV 检测。
本研究CNV-Seq 检出一例t(8;18)(q21;q23) 相互易位样本的8号染色体末端0.9 Mb 的缺失,这一微小 CNV 由 45 个连续测序单元中的累积测序片段得来,其对应CNV 终点为 ±20 kb。近期,一项通过 CNV-Seq 检测 200 个胚胎样品WGA产物的有效性研究显示 [17],CNV-Seq对染色体整倍体及非整倍体同样具有高度的灵敏度和特异性,仅存在一例 18 号染色体三体假阳性。该研究将测序片段分配到1 Mb的测序单元中,从而将CNV的检测范围降至 14Mb、17Mb及20 Mb。然而,使用较大测序单元(1 Mb)将会影响检测分辨率,因而该数据分析方法[17]可能无法对临床意义较小的胚胎 CNVs 实现精确的可重复性检测。
值得注意的是,本研究Array CGH 和 CNV-Seq 仅对一例t(8;18)(q21;q23) 易位患者的检测结果不一致。CNV-Seq 明确检出此例样本的两处染色体非平衡易位以及一处Xp 缺失,而 Array CGH 完全漏检上述异常。分析该样本相应的 Array CGH结果,发现由于 WGA 反应效果不佳,从而全基因组扩增出现偏差,导致Array CGH结果存在显著的背景杂交噪声。CNV-Seq 可对密度明显较高的测序片段进行分析以获取更高的分辨率,故能克服基因组偏差产生的局部影响。因而即使这些偏差因 WGA 反应不佳而扩大,CNV-Seq 仍能检测胚胎的染色体异常[29,30]。
相较于近期报道的高通量测序方法[17],CNV-Seq 的另一个优点是能够精确定量CNV。本研究所有样本的CNV拷贝数均在相应理论值(分别为 3 和 1)的20% 范围内。熟练运用 CNV-Seq,准确绘制微小 CNV 的精确图谱,可将CNV-Seq的临床应用从染色体非整倍体扩展至某些微小缺失引起的单基因疾病[31]。在此前提下,CNV-Seq 检出的任何微小 CNV 均可绘制到分辨率为 20 kb的测序单元中,从而与 hg19参比基因组对比,识别CNV与某项疾病综合征之间的关联。更进一步,CNV-Seq 可定量分析 X 和 Y 染色体,对提高性染色体非整倍体的检测准确度极其重要[2,32]。临床 PGD 较多使用滋养外胚层活检样品[8,10,33],而这些样品中可能存在染色体嵌合[34],使用CNV-Seq 从低水平或中等水平嵌合体中检出正常样品,对减少误诊率至关重要。
虽然微阵列技术已在 PGD 运用中显示较高的可靠性和准确性[2,35],但这项技术费时且流程繁琐(长达 24 小时),同时微阵列玻片及 DNA 标记、杂交试剂费用高昂。此外,微阵列技术的样本通量可扩展性小,任意一天内获取的样品量都可能少于或多于一张微阵列芯片所能承载的最大样本量。与之相对,在工作繁忙的 PGD 实验室应用 CNV-Seq 具有多个优点。首先,从WGA 到最终检测,CNV-Seq 工作流程简单,利用 Illumina HiSeq 2500 测序平台可在约 16 小时内完成检测,有望移植新鲜胚胎。其次,CNV-Seq 可对1~100 个样品进行灵活检测,能批量处理每日样本。再次,基于WGA 、文库构建、HiSeq 2500平台运行等费用,以每次运行的最大样本量进行成本计算,平均到单个样本的检测费用仅为当前微阵列芯片平台的1/5~1/2。再次,CNV-Seq 标准流程包含最少次数的胚胎移植,每个样品的所有测序片段均引入序列标签,能显著减少人为错误以及由此引发的误诊。最后,测序试剂可能会随着时间推移而持续下降,若检测成本下降可惠及患者,则以CNV-Seq为代表的NGS技术可成为 PGD 实验室及患者可负担的检测项目。
综上所述,本研究验证了CNV-Seq 检测染色体非平衡易位的性能。结合近期报道的另外两项研究[17,18],推测随着NGS 技术的迅速发展,有望取代 Array CGH 及 SNP array对胚胎染色体异常进行 PGD检测。若提高测序深度,CNV-Seq 的临床应用则可能使从染色体非整倍体扩展至已知染色体微小 CNV的准确检测上[36]。重要的是,NGS 工作流程简单、劳动强度小、试剂消耗更加经济,可提高实验室PGD 检测的总体效率,最终使更多患者受益。在科研水平,CNV-Seq 可用于全面评估人类胚胎染色体异常,并帮助认识配子形成及引起胚胎植入前染色体不稳定的关键机制。
参考文献:
1. Hardy K, Handyside AH. Biopsy of cleavage stage human embryos and diagnosis of single gene defects by DNA amplification. Arch Pathol Lab Med 1992; 116:388–392.
2. Munne S. Preimplantation genetic diagnosis for aneuploidy and translocations using array comparative genomic hybridization. Curr Genomics 2012; 13:463–470.
3. Munne S, Weier HU. Simultaneous enumeration of chromosomes 13, 18, 21, X, and Y in interphase cells for preimplantation genetic diagnosis of aneuploidy. Cytogenet Cell Genet 1996; 75:263–270.
4. Munne S, Sandalinas M, Escudero T, Fung J, Gianaroli L, Cohen J. Outcome of preimplantation genetic diagnosis of translocations. Fertil Steril 2000; 73:1209–1218.
5. Telenius H, Carter NP, Bebb CE, Nordenskjold M, Ponder BA, Tunnacliffe A. Degenerate oligonucleotide-primed PCR: general amplification of target DNA by a single degenerate primer. Genomics 1992; 13: 718–725.
6. Wilton L, Williamson R, McBain J, Edgar D, Voullaire L. Birth of a healthy infant after preimplantation confirmation of euploidy by comparative genomic hybridization. N Engl J Med 2001; 345:1537–1541.
7. Fiorentino F, Spizzichino L, Bono S, Biricik A, Kokkali G, Rienzi L, Ubaldi FM, Iammarrone E, Gordon A, Pantos K. PGD for reciprocal and Robertsonian translocations using array comparative genomic hybridization. Hum Reprod 2011; 26:1925–1935.
8. Schoolcraft WB, Treff NR, Stevens JM, Ferry K, Katz-Jaffe M, Scott RT Jr. Live birth outcome with trophectoderm biopsy, blastocyst vitrification, and single-nucleotide polymorphism microarray-based comprehensive chromosome screening in infertile patients. Fertil Steril 2011; 96:638–640.
9. Tan YQ, Tan K, Zhang SP, Gong F, Cheng DH, Xiong B, Lu CF, Tang XC, Luo KL, Lin G, Lu GX. Single-nucleotide polymorphism microarraybased preimplantation genetic diagnosis is likely to improve the clinical outcome for translocation carriers. Hum Reprod 2013; 28:2581–2592.
10. Treff NR, Tao X, Ferry KM, Su J, Taylor D, Scott RT Jr. Development and validation of an accurate quantitative real-time polymerase chain reaction-based assay for human blastocyst comprehensive chromosomal aneuploidy screening. Fertil Steril 2012; 97:819–824.
11. Kuliev A, Zlatopolsky Z, Kirillova I, Spivakova J, Cieslak Janzen J. Meiosis errors in over 20,000 oocytes studied in the practice of preimplantation aneuploidy testing. Reprod Biomed Online 2011; 22:2–8.
12. Voet T, Vanneste E, Van der Aa N, Melotte C, Jackmaert S, Vandendael T, Declercq M, Debrock S, Fryns JP, Moreau Y, D’Hooghe T, Vermeesch JR. Breakage-fusion-bridge cycles leading to inv dup del occur in human cleavage stage embryos. Hum Mutat 2011; 32:783–793.
13. Voet T, Vanneste E, Vermeesch JR. The human cleavage stage embryo is a cradle of chromosomal rearrangements. Cytogenet Genome Res 2011; 133:160–168.
14. Vanneste E, Voet T, Le Caignec C, Ampe M, Konings P, Melotte C, Debrock S, Amyere M, Vikkula M, Schuit F, Fryns JP, Verbeke G, et al. Chromosome instability is common in human cleavage-stage embryos.
Nat Med 2009; 15:577–583.
15. Hou Y, Fan W, Yan L, Li R, Lian Y, Huang J, Li J, Xu L, Tang F, Xie XS, Qiao J. Genome analyses of single human oocytes. Cell 2013; 155: 1492–1506.
16. Wang L, Wang X, Zhang J, Song Z, Wang S, Gao Y, Wang J, Luo Y, Niu Z, Yue X, Xu G, Cram DS, et al. Detection of chromosomal aneuploidy in human preimplantation embryos by next-generation sequencing. Biol Reprod 2014; 90:95.
17. Fiorentino F, Biricik A, Bono S, Spizzichino L, Cotroneo E, Cottone G, Kokocinski F, Michel CE. Development and validation of a nextgeneration sequencing-based protocol for 24-chromosome aneuploidy screening of embryos. Fertil Steril 2014; 101:1375–1382.
18. Yin X, Tan K, Vajta G, Jiang H, Tan Y, Zhang C, Chen F, Chen S, Pan X, Gong C, Li X, Lin C, et al. Massively parallel sequencing for chromosomal abnormality testing in trophectoderm cells of human blastocysts. Biol Reprod 2013; 88:69.
19. Song Y, Liu C, Qi H, Zhang Y, Bian X, Liu J. Noninvasive prenatal testing of fetal aneuploidies by massively parallel sequencing in a prospective Chinese population. Prenat Diagn 2013; 33:700–706.
20. Wang Y, Chen Y, Tian F, Zhang J, Song Z, Wu Y, Han X, Hu W, Ma D, Cram D, Cheng W. Maternal mosaicism is a significant contributor to discordant sex shromosomal aneuploidies associated with noninvasive prenatal testing. Clin Chem 2013; 60:251–259.
21. Wang Y, Zhu J, Chen Y, Lu S, Chen B, Zhao X, Wu Y, Han X, Ma D, Liu Z, Cram D, Cheng W. Two cases of placental T21 mosaicism: challenging the detection limits of non-invasive prenatal testing. Prenat Diagn 2013; 33:1207–1210.
22. Mao J, Wang T, Wang BJ, Liu YH, Li H, Zhang J, Cram D, Chen Y. Confined placental origin of the circulating cell free fetal DNA revealed by a discordant non-invasive prenatal test result in a trisomy 18 pregnancy. Clin Chim Acta 2014; 433:190–193.
23. Yu C, Wang L, Li J, Guo C, Guo X, Zhang X, Xu Y, Yao Y. Ovarian stimulation reduces IL-6 release from mouse and human pre-implantation embryos. Am J Reprod Immunol 2012; 68:199–204.
24. Shen J, Cram DS, Wu W, Cai L, Yang X, Sun X, Cui Y, Liu J. Successful PGD for late infantile neuronal ceroid lipofuscinosis achieved by combined chromosome and TPP1 gene analysis. Reprod Biomed Online 2013; 27:176–183.
25. Liang D, Lv W, Wang H, Xu L, Liu J, Li H, Hu L, Peng Y, Wu L. Noninvasive prenatal testing of fetal whole chromosome aneuploidy by massively parallel sequencing. Prenat Diagn 2013; 33:409–415.
26. Li H, Durbin R. Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics 2009; 25:1754–1760.
27. Hocking TD, Schleiermacher G, Janoueix-Lerosey I, Boeva V, Cappo J, Delattre O, Bach F, Vert JP. Learning smoothing models of copy number profiles using breakpoint annotations. BMC Bioinformatics 2013; 14:164.
28. Picard F, Robin S, Lavielle M, Vaisse C, Daudin JJ. A statistical approach for array CGH data analysis. BMC Bioinformatics 2005; 6:27.
29. Treff NR, Su J, Tao X, Northrop LE, Scott RT, Jr. Single-cell wholegenome amplification technique impacts the accuracy of SNP microarraybased genotyping and copy number analyses. Mol Hum Reprod 2011; 17: 335–343.
30. Van der Aa N, Esteki MZ, Vermeesch JR, Voet T. Preimplantation genetic diagnosis guided by single-cell genomics. Genome Med 2013; 5:71.
31. Stankiewicz P, Lupski JR. Structural variation in the human genome and its role in disease. Annu Rev Med 2010; 61:437–455.
32. Simpson JL, Samango-Sprouse C. Prenatal diagnosis and 47,XXY. Am J Med Genet C Semin Med Genet 2013; 163C:64–70.
33. Schoolcraft WB, Katz-Jaffe MG. Comprehensive chromosome screening of trophectoderm with vitrification facilitates elective single-embryo transfer for infertile women with advanced maternal age. Fertil Steril 2013; 100:615–619.
34. Liu J, Wang W, Sun X, Liu L, Jin H, Li M, Witz C, Williams D, Griffith J, Skorupski J, Haddad G, Gill J. DNA microarray reveals that high proportions of human blastocysts from women of advanced maternal age are aneuploid and mosaic. Biol Reprod 2012; 87:148.
35. Handyside AH. 24-chromosome copy number analysis: a comparison of available technologies. Fertil Steril 2013; 100:595–602.
36. Schaaf CP, Wiszniewska J, Beaudet AL. Copy number and SNP arrays in clinical diagnostics. Annu Rev Genomics Hum Genet 2011; 12:25–51.