上皮性卵巢癌PARP抑制剂相关生物标志物检测的中国专家共识
中国抗癌协会妇科肿瘤专业委员会,中华医学会病理学分会
关键词:上皮性卵巢癌;PARP抑制剂;标志物;检测
共识执笔人:
温 灏 复旦大学附属肿瘤医院
吴焕文 中国医学科学院北京协和医院
共识专家组组长:
吴小华 复旦大学附属肿瘤医院
梁智勇 中国医学科学院北京协和医院
共识专家组成员(按姓氏笔画排序):
王 莉 河南省肿瘤医院
王丹波 辽宁省肿瘤医院
王 静 湖南省肿瘤医院
尹如铁 四川大学华西第二医院
叶 庆 中国科学技术大学附属第一医院
朱笕青 中国科学院大学附属肿瘤医院
刘继红 中山大学肿瘤防治中心
李 力 广西医科大学附属肿瘤医院
杨宏英 云南省肿瘤医院
吴令英 中国医学科学院肿瘤医院
张师前 山东大学齐鲁医院
张智弘 江苏省人民医院
邵建永 中山大学肿瘤防治中心
林仲秋 中山大学孙逸仙纪念医院
欧阳能太 中山大学孙逸仙纪念医院
周 琦 重庆大学附属肿瘤医院
周晓燕 复旦大学附属肿瘤医院
高雨农 北京大学肿瘤医院
盛修贵 中国医学科学院肿瘤医院深圳医院
卵巢癌是女性生殖系统常见的恶性肿瘤,中国每年卵巢癌新发病例为52 100例,死亡病例达22 500例[1]。卵巢癌的发病风险因素包括家族史、遗传因素、年龄、体质量、子宫内膜异位症、未生育、激素替代治疗等。由于缺乏有效的早期筛查手段,患者就诊时多为晚期,在中国卵巢癌患者的5年生存率约为40%[2]。近年来,随着聚腺苷二磷酸核糖聚合酶[poly(ADP-ribose)polymerase,PARP]抑制剂广泛应用于临床,有效地延长了晚期卵巢癌患者的无进展生存期(progression-free survival,PFS),改变了卵巢癌的治疗格局。对卵巢癌患者进行相关的生物标志物检测,有助于指导临床合理用药,改善卵巢癌患者的治疗结局。为规范卵巢癌PARP抑制剂相关的生物标志物检测,中国抗癌协会妇科肿瘤专业委员会与中华医学会病理学分会联合制定本专家共识。
本共识采用以下推荐级别(表1),相关推荐同样适用于输卵管癌及原发性腹膜癌。

1卵巢癌与同源重组修复缺陷
约50%的上皮性卵巢癌存在同源重组修复缺陷(homologous recombination deficiency,HRD)。同源重组修复(homologous recombination repair,HRR)是正常细胞修复DNA双链断裂损伤(double strand break,DSB)的重要途径,HRD导致细胞DNA双链断裂损伤修复途径缺陷,表现为对引起DNA断裂的铂类药物以及PARP抑制剂高度敏感,因而HRD已成为卵巢癌治疗相关的重要生物标志物。HRR通路相关的基因突变是导致HRD的主要原因,卵巢癌中常见的HRR突变有BRCA1、BRCA2、ATM、BARD1、BRIP1、CHEK1、CHEK2、FAM175A、MRE11A、NBN、PALB2、RAD51C、RAD51D等,其中BRCA1和BRCA2突变较为常见,在上皮性卵巢癌中胚系BRCA1/2突变占14%~15%,在高级别浆液性卵巢癌中BRCA1/2突变更加常见,约22.6%存在胚系BRCA1/2突变,6%~7%存在体细胞BRCA1/2突变,BRCA1的突变频率高于BRCA2。除突变外,基因也可以通过表观遗传学机制失活,高级别浆液性卵巢癌中约10%存在BRCA1基因启动子的甲基化,约2%存在RAD51C基因启动子的甲基化[3-4]。目前,仍有一部分卵巢癌发生HRD的机制尚不明确。
2PARP抑制剂相关的生物标志物
近年来,多项PARP抑制剂的临床研究证实,其对于卵巢癌患者的显著疗效,改变了卵巢癌患者的诊疗策略。目前,与PARP抑制剂治疗相关的生物标志物主要有BRCA1/2基因突变、HRR基因突变、BRCA1/RAD51C启动子甲基化、HRD状态等。
2.1 BRCA1/2基因突变
BRCA1/2是重要的抑癌基因,对于维持细胞正常的生长增殖至关重要,也是维持细胞HRR功能最重要的基因。携带BRCA1/2突变的多种肿瘤对PARP抑制剂敏感,在SOLO-1研究中,携带胚系或体细胞BRCA1/2突变的晚期上皮性卵巢癌患者在初始治疗缓解后应用奥拉帕利维持治疗,相比安慰剂,患者复发或死亡风险下降70%,中位PFS延长3年以上[5]。
BRCA1/2胚系突变还与肿瘤的遗传易感性相关。携带有BRCA1/2胚系致病性变异的女性,乳腺癌发生风险提高5倍,卵巢癌发生风险提高10~30倍[6-8],此外,前列腺癌、胰腺癌、男性乳腺癌、恶性黑色素瘤等的发病风险也会显著增高[9-12]。明确卵巢癌患者的BRCA1/2胚系突变状态,有助于对患者及其家系进行遗传风险管理,包括家系验证、制定筛查方案、化学预防、预防性手术、生殖干预等[13-14]。
2.2 HRR基因突变
HRR是一个复杂的生物学过程,开始于细胞内DNA损伤感应蛋白质对于DSB的识别,这个过程主要依赖于MRN(MRE11、RAD50、NBS1)蛋白复合物,随后在DNA酶的作用下由5’到3’对DNA进行切割,ATM、RPA等蛋白结合于突出的单链DNA阻止其进一步降解;以RAD51为核心的DNA重组酶随后结合于单链DNA并在姐妹染色单体上寻找同源序列以作为后续DNA修复的模板,在这个过程中,RAD51借由BRCA2募集到RPA结合的单链DNA上,而BRCA2的募集则依赖于BRCA1和PALB2[15-16]。体外实验表明,除BRCA1/2外,其他HRR基因突变也可能导致细胞对PARP抑制剂敏感[17],在几项卵巢癌PARP抑制剂相关的临床研究中,同样得到了证实(表2)[18-21]。
2.3 BRCA1/RAD51C启动子甲基化
基因的表观遗传学变化同样可能引起细胞发生HRD,卵巢癌中较常见的是BRCA1和RAD51C的启动子甲基化,BRCA1或RAD51C的甲基化将导致对应的基因表达下调,并且通常与BRCA1/2或其他HRR相关基因的变异互斥[22]。在患者来源异种移植瘤(patient-derived xenograft,PDX)小鼠模型、卵巢癌细胞系及PARP抑制剂的临床研究中发现,BRCA1或RAD51C甲基化与rucaparib的敏感性相关,纯合BRCA1甲基化对rucaparib高度敏感,而杂合BRCA1甲基化对rucaparib表现出耐药。对ARIEL2研究的数据分析表明,携带纯合BRCA1甲基化与携带BRCA1/2突变的卵巢癌患者接受rucaparib单药治疗的中位PFS接近(14.5个月 vs 12.8个月)[23-24]。研究发现BRCA1的甲基化也可能与患者对奥拉帕利的长期获益相关[25],但目前临床研究证据仍然较少,BRCA1/RAD51C甲基化检测目前主要用于了解卵巢癌的HRD状态。
2.4 HRD状态
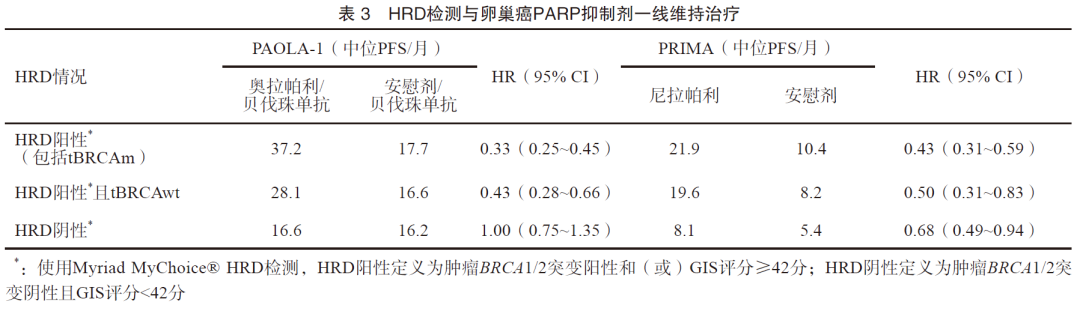
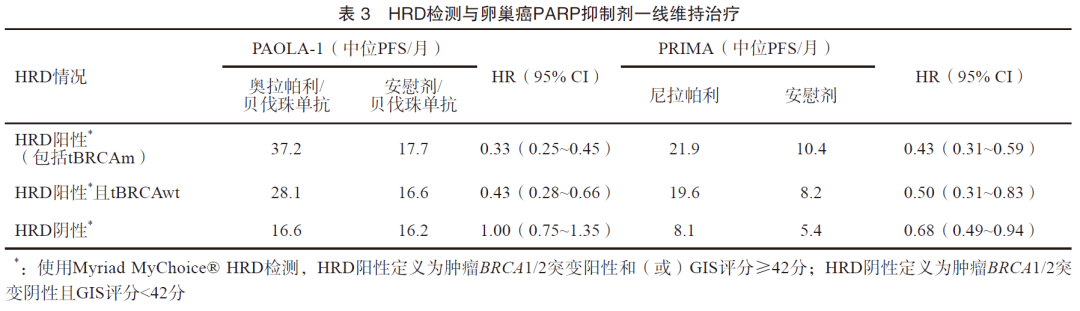
在新诊断的卵巢癌中,BRCA1/2和HRD检测被推荐用于指导一线卵巢癌的治疗方案选择[26-28]。PAOLA-1研究表明,针对一线含铂类药物联合贝伐珠单抗治疗有效的上皮性卵巢癌患者,继续使用贝伐珠单抗的同时联合或不联合奥拉帕利进行维持治疗,在HRD阳性的患者中,奥拉帕利联合贝伐珠单抗组患者中位PFS延长19.5个月(37.2个月 vs 17.7个月),复发或死亡风险降低67%。即使在BRCA1/2野生型HRD阳性的患者中,奥拉帕利联合贝伐珠单抗组患者中位PFS也可延长11.5个月(28.1个月 vs 16.6个月),复发或死亡风险降低57%(表3)[29]。PRIMA研究表明,在初始治疗缓解后应用尼拉帕利对比安慰剂进行维持治疗能够显著获益,亚组分析结果表明,HRD阳性患者的获益(21.9个月 vs 10.4个月)优于HRD阴性患者(8.1个月 vs 5.4个月)(表3)[30]。
除此之外,包括突变特征Signature 3、HRDetect、功能性HRD检测(RAD51 Foci)等在内的新兴生物标志物及检测方法对于PARP抑制剂敏感性的预测也在研究之中[31-33]。
3PARP抑制剂相关的生物标志物检测方法
3.1 BRCA1/2检测
BRCA1/2突变分为胚系突变和体细胞突变两种。胚系BRCA1/2突变起源于生殖细胞,存在于机体的每一个细胞中;体细胞BRCA1/2突变仅存在于肿瘤细胞中。肿瘤组织检测可同时获得胚系及体细胞BRCA1/2的突变信息,对于突变检测阳性的患者建议进一步行胚系突变分析,以区分胚系或体细胞突变。肿瘤检测一般使用手术或穿刺获得的肿瘤组织样本,胚系检测一般使用血液、唾液、口腔拭子等样本,目前以血液为主[34]。BRCA1/2变异类型多样,且遍布于基因全长。国内对于BRCA1/2检测一般采用二代测序(next generation sequencing,NGS)或称高通量测序的方法。依据胚系BRCA1/2变异的解读原则,将胚系BRCA1/2基因变异按照风险程度由高至低分为5类:致病性(5类)、可能致病性(4类)、意义未明(3类)、可能良性(2类)和良性(1类)。其中,BRCA1/2致病性和可能致病性的变异通常被称为BRCA1/2基因突变阳性[35-37]。对于体细胞BRCA1/2变异的解读,一般参考肿瘤变异的解读原则,关注该变异对临床实践的影响,如对某种治疗的敏感性、耐药性的预测,对疾病的诊断或预后的影响等[38-39]。
3.2 HRR基因检测
与BRCA1/2基因检测类似,HRR基因检测同样采用NGS方法,通常在多基因panel上进行。HRR突变同样分为胚系变异和体细胞变异,解读原则与BRCA1/2相同。不同的HRR基因突变对于PARP抑制剂的敏感性可能不同,况且目前在卵巢癌中的研究证据有限,因此对于HRR基因突变临床意义的解读需要谨慎。
3.3 HRD检测
HRD检测并无统一标准,其原理是基于细胞内因HRD而引起的DNA损伤,将以一些特定且可识别的方式在基因组上留下痕迹,如杂合性丢失(loss of heterozygosity,LOH)、端粒等位基因失平衡(telomeric allelic imbalance,TAI)和大片段迁移(large-scale state transitions,LST)等[40-43]。HRD检测采用NGS方法,通常包括两个部分,BRCA1/2突变状态及基因组不稳定性状态的评分(genomic instability score,GIS),或称HRD评分(HRD score)。对于后者,一般通过对细胞内单核苷酸多态性位点(single nucleotide polymorphism,SNP)进行检测和计算得出。
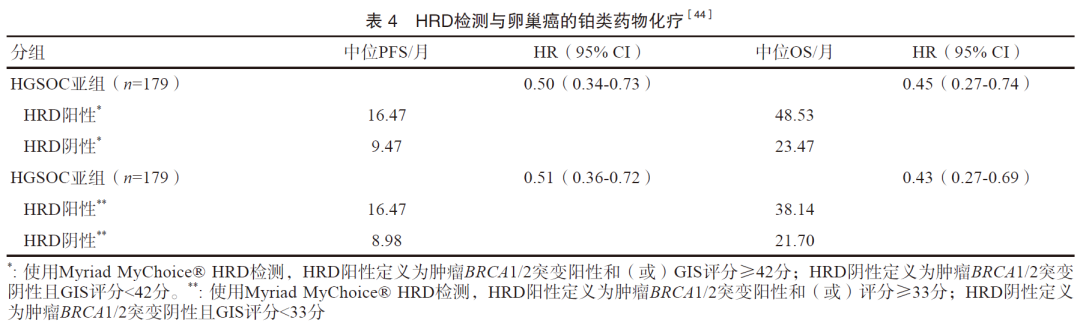
目前,全球范围内仅2种HRD检测产品在大型Ⅲ期临床研究中得到验证,并已经得到美国食品药品管理局(Food and Drug Administration,FDA)的批准:Myriad myChoice® CDx(Myriad Genetic Laboratories, Inc.)和FoundationFocusTM CDx BRCA LOH(Foundation Medicine, Inc.)。在Myriad MyChoice® CDx检测中,HRD阳性定义为肿瘤BRCA1/2突变和(或)GIS评分≥42分,GIS评分由LOH、TAI、LST三项综合计算得出,阈值的设定基于BRCA缺陷的卵巢癌和乳腺癌肿瘤样本第5分位的HRD分值,并最早在乳腺癌和卵巢癌对含铂类药物化疗敏感性的预测中被证实有效(表4)。在FoundationFocusTM CDx BRCA LOH检测中,HRD阳性定义为肿瘤BRCA1/2突变和(或)基因组LOH评分≥16%,阈值的设定最初基于其对卵巢癌患者接受含铂类药物化疗效果的有效区分,随后根据其对卵巢癌患者接受rucaparib治疗效果的区分进行了调整[44-46]。
国内HRD检测产品正在研发过程中,根据国家药品监督管理局(National Medical Products Administration,NMPA)及美国FDA对于伴随诊断试剂、基于NGS技术的肿瘤基因突变检测试剂等通用的技术指导原则,体外诊断试剂盒通常需要经过设计(design)、开发(development)、分析性能验证(analytical validation)、临床验证(clinical validation)几个阶段。体外诊断试剂的设计开发阶段需考虑产品的预期用途、使用人群、检测样本、检测方法及需达到的技术指标等。分析性能验证的主要目标是评估产品的有效性和安全性,通过一组预定义的性能评价方式,以证明性能是否满足其预期用途并符合预定义的性能标准,如准确度(accuracy)、精密度(precision)、检测限(limit of detection,LoD)、分析特异性(analytical specificity)等。临床验证的主要目标是评估产品的临床性能是否满足预期用途,如是否能鉴别出可获益于某种治疗方式的人群等[47-53]。
目前国内尚无HRD试剂盒的技术指导原则,基于HRD检测对于指导卵巢癌患者使用PARP抑制剂治疗的临床意义,以及国内外对于体外诊断试剂盒的相关指导原则,经过专家组讨论,对国内HRD试剂盒的临床验证及性能提出如下建议:
⑴ 对于HRD检测的临床验证,优先推荐在有严格随访计划的随机对照或单臂的干预性临床研究,或前瞻性队列研究中进行验证(1类),在以上数据较难获取的前提下,可选择在回顾性队列或真实世界研究中进行初步验证(2A类)。
⑵ 对于HRD检测的临床性能,阳性患者接受PARP抑制剂或铂类药物化疗的获益应与历史数据具有可比性,具体如下(表5):对于新诊断的卵巢癌患者,使用PARP抑制剂单药或联合方案进行一线化疗后的维持治疗,HRD阳性人群PFS与历史数据具有可比性;HRD阳性人群使用PARP抑制剂对比安慰剂维持治疗PFS的HR与历史数据具有可比性;HRD阳性人群对比HRD阴性人群,使用PARP抑制剂进行维持治疗PFS的HR与历史数据具有可比性(1类)。

在以上数据较难获取的前提下,可选择在以下人群中进行初步验证:
① 对于使用铂类药物进行一线化疗而未接受维持治疗的卵巢癌患者,HRD阳性或阴性人群化疗PFS与历史数据具有可比性;HRD阳性人群对比HRD阴性人群,化疗PFS的HR与历史数据具有可比性(2A类)。② 对于铂敏感复发的卵巢癌患者,使用PARP抑制剂进行化疗后的维持治疗,HRD阳性或阴性人群使用PARP抑制剂进行维持治疗,PFS与历史数据具有可比性;HRD阳性人群对比HRD阴性人群,使用PARP抑制剂进行维持治疗PFS的HR与历史数据具有可比性(2A类)。③ 对于既往接受过2线或以上含铂类药物化疗的铂敏感复发性卵巢癌患者,HRD阳性人群使用PARP抑制剂单药进行治疗,ORR或PFS与历史数据具有可比性(2A类)。
4对于卵巢癌患者PARP抑制剂生物标志物检测的建议
国内外权威指南推荐卵巢癌患者在初次病理学检查确诊时,需要明确肿瘤的BRCA1/2突变状态,以指导后续维持治疗;HRD状态对于肿瘤BRCA1/2检测阴性患者维持治疗的选择具有重要的参考价值[26-28,54-56]。基于国内外指南及共识的推荐、PARP抑制剂国内外药物适应证,以及中国卵巢癌患者临床诊疗现状,经过专家组讨论,对中国卵巢癌患者PARP抑制剂相关的生物标志物检测进行如下推荐:
⑴ 推荐所有非黏液性卵巢癌患者在初次病理学检查确诊时,明确肿瘤BRCA1/2的突变(包括胚系和体细胞突变)状态,对于Ⅰ期患者仅需明确胚系BRCA1/2突变状态(1类)。① 如果患者仅行肿瘤组织BRCA1/2检测,且突变状态为阳性,建议进一步采用血液或唾液样本进行BRCA1/2胚系检测,以明确该变异是否为胚系变异(1类)。② 如果患者仅行肿瘤组织BRCA1/2检测,且突变状态为阴性,建议进一步对血液样本进行大片段重排(large genomic rearrangement,LGR)变异的检测以明确是否存在BRCA1/2的胚系LGR变异(1类)。③ 如患者仅行胚系BRCA1/2检测,且结果为阳性,则无需再对肿瘤组织进行BRCA1/2检测(1类)。④ 如患者仅行胚系BRCA1/2检测(包含针对LGR的MLPA检测),且结果为阴性,则需要对肿瘤组织进行BRCA1/2检测(1类)。⑤ 根据当地检测策略并综合成本效益,可以同时进行BRCA1/2的胚系及肿瘤突变检测,或胚系、肿瘤检测序贯进行(2A类)。
⑵ 对于新诊断的晚期卵巢癌患者(目前主要证据在高级别浆液性卵巢癌和高级别子宫内膜样癌),HRD状态(包括BRCA1/2和HRD score)有助于医师选择不同维持治疗方案以期达到最佳治疗效果:
① 建议进行HRD检测(包括BRCA1/2和HRD score)(2A类);如患者存在抗血管生成抑制剂治疗的禁忌证,或不考虑抗血管生成抑制剂治疗时,HRD状态对于维持治疗的疗效预测及预后判断仍有参考价值(2B类)。② 如既往接受过肿瘤BRCA1/2检测,且结果为阳性,不需要再补充进行HRD检测(1类)。③ 如既往接受过肿瘤BRCA1/2检测,且结果为阴性,建议对肿瘤样本进行HRD检测以明确是否为HRD阳性(2A类)。④ 当HRD检测不可及时,可考虑对肿瘤组织进行HRR基因检测(3类)。
⑶ 对于铂敏感复发的卵巢癌患者,BRCA1/2突变状态及HRD状态并不作为含铂类药物化疗后PARP抑制剂维持治疗的选择标准,但对于患者疗效预测及预后判断具有一定的参考价值:
① 既往已经接受过BRCA1/2或HRD的检测,即便有更新的肿瘤样本可及时,也暂不推荐对同一检测项目重复进行检测(2A类)。② 可考虑进行HRD检测(包括BRCA1/2和HRD score)(2B类)。③ 如既往接受过肿瘤BRCA1/2检测,且结果为阳性,不需要补充HRD检测(1类)。④ 如既往接受过肿瘤BRCA1/2检测,且结果为阴性,可考虑进行HRD检测(2B类)。⑤ 当HRD检测不可及时,可考虑进行肿瘤组织HRR基因检测(3类)。
⑷ 对于考虑使用PARP抑制剂作为单药挽救性治疗的后线卵巢癌患者:
① 既往已经接受过BRCA1/2或HRD的检测,即便有更新的肿瘤样本可及时,也暂不推荐对同一检测项目重复进行检测(2B类)。② 铂敏感复发的患者,推荐进行HRD检测(包括BRCA1/2和HRD score)(2A类)。③ 铂耐药复发的患者,仅需接受胚系和(或)肿瘤BRCA1/2检测(2A类)。④ 当HRD检测不可及时,可考虑进行肿瘤组织HRR基因检测(3类)。
约25%的上皮性卵巢癌发病与遗传因素相关,推荐上皮性卵巢癌患者接受遗传风险评估,相关遗传咨询、风险评估及检测建议参考其他相关指南[28,34,56]。
参考文献
CHEN W, ZHENG R, BAADE P D, et al. Cancer statistics in China, 2015[J]. CA Cancer J Clin, 2016, 66(2): 115-132.
JIANG X, TANG H J, CHEN T. Epidemiology of gynecologic cancers in China[J]. J Gynecol Oncol, 2018, 29(1): e7.
KONSTANTINOPOULOS P A, CECCALDI R, SHAPIRO G I, et al. Homologous recombination deficiency: exploiting the fundamental vulnerability of ovarian cancer[J]. Cancer Discov, 2015, 5(11): 1137-1154.
PENNINGTON K P, WALSH T, HARRELL M I, et al. Germline and somatic mutations in homologous recombination genes predict platinum response and survival in ovarian, fallopian tube, and peritoneal carcinomas[J]. Clin Cancer Res, 2014, 20(3): 764-775.
MOORE K, COLOMBO N, SCAMBIA G, et al. Maintenance olaparib in patients with newly diagnosed advanced ovarian cancer[J]. N Engl J Med, 379, 26(2018): 2495-2505.
ANTONIOU A, PHAROAH P D, NAROD S, et al. Average risks of breast and ovarian cancer associated with BRCA1 or BRCA2 mutations detected in case series unselected for family history: a combined analysis of 22 studies[J]. Am J Hum Genet, 2003, 72(5): 1117-1130.
CHEN S, PARMIGIANI G. Meta-analysis of BRCA1 and BRCA2 penetrance[J]. J Clin Oncol, 2007, 25(11): 1329-1333.
HOWLADER N, NOONE A M, KRAPCHO M, et al. SEER cancer statistics review, 1975-2011. Bethesda, MD: National Cancer Institute, 2014, 19[EB/OL]. https://seer.cancer.gov/archive/csr/1975_2011/.
LEONGAMORNLERT D, MAHMUD N, TYMRAKIEWICZ M, et al. Germline BRCA1 mutations increase prostate cancer risk[J]. Br J Cancer, 2012, 106(10): 1697-1701.
IQBAL J, RAGONE A, LUBINSKI J, et al. The incidence of pancreatic cancer in BRCA1 and BRCA2 mutation carriers[J]. Br J Cancer, 2012, 107(12): 2005-2009.
TAI Y C, DOMCHEK S, PARMIGIANI G, et al. Breast cancer risk among male BRCA1 and BRCA2 mutation carriers[J]. J Natl Cancer Inst, 2007, 99(23): 1811-1814.
MORAN A, O’HARA C, KHAN S, et al. Risk of cancer other than breast or ovarian in individuals with BRCA1 and BRCA2 mutations[J]. Fam Cancer, 2012, 11(2): 235-242.
MEIJERS-HEIJBOER H, VAN GEEL B, VAN PUTTEN W L, et al. Breast cancer after prophylactic bilateral mastectomy in women with a BRCA1 or BRCA2 mutation[J]. N Engl J Med, 2001, 345(3): 159-164.
DOMCHEK S M, FRIEBEL T M, SINGER C F, et al. Association of risk-reducing surgery in BRCA1 or BRCA2 mutation carriers with cancer risk and mortality[J]. JAMA, 2010, 304(9): 967-975.
LORD C J, ASHWORTH A. BRCAness revisited[J]. Nat Rev Cancer, 2016, 16(2): 110.
SUN Y, MCCORVIE T J, YATES L A, et al. Structural basis of homologous recombination[J]. Cell Mol Life Sci, 2020, 77(1): 3-18.
MCCABE N, TURNER N C, LORD C J, et al. Deficiency in the repair of DNA damage by homologous recombination and sensitivity to poly (ADP-ribose) polymerase inhibition[J]. Cancer Res, 2006, 66(16): 8109-8115.
HOPPE M M, SUNDAR R, TAN D S P, et al. Biomarkers for homologous recombination deficiency in cancer[J]. J Natl Cancer Inst, 2018, 110(7): 704-713.
HODGSON D R, DOUGHERTY B A, LAI Z, et al. Candidate biomarkers of PARP inhibitor sensitivity in ovarian cancer beyond the BRCA genes[J]. Brit J Cancer, 2018, 119(11): 1401-1409.
MIRZA M R, FENG B, SHAN M, et al. Elucidation of PARP inhibitor activity in BRCAwt recurrent ovarian cancer by HRR mutational gene profile analysis[J]. J Clin Oncol, 2019, 37(15): 5568.
O’MALLEY, DAVID M. Post progression outcomes in patients with ovarian carcinoma associated with a mutation in a non-BRCA homologous recombination repair gene receiving rucaparib maintenance treatment: results from the phase 3 study ARIEL3[C]. SGO 2020 Annual Meeting on Women’s Cancer. SGO, 2020.
MOSCHETTA M, GEORGE A, KAYE S B, et al. BRCA somatic mutations and epigenetic BRCA modifications in serous ovarian cancer[J]. Ann Oncol, 2016, 27(8): 1449-1455.
SWISHER E M, HARRELL M I, LIN K, et al. BRCA1 and RAD51C promoter hypermethylation confer sensitivity to the PARP inhibitor rucaparib in patients with relapsed, platinum-sensitive ovarian carcinoma in ARIEL2 Part 1[J]. Gynecol Oncol, 2017, 145: 5.
KONDRASHOVA O, TOPP M, NESIC K, et al. Methylation of all BRCA1 copies predicts response to the PARP inhibitor rucaparib in ovarian carcinoma[J]. Nat Commun, 2018, 9(1): 1-16.
FRANZESE E, CENTONZE S, DIANA A, et al. Genomic profile and BRCA1 promoter methylation status in BRCA mutated ovarian cancer: new insights in predictive biomarkers of olaparib response[J]. Front Oncol, 2019, 9: 1289.
中华医学会妇科肿瘤学分会. 卵巢癌PARP抑制剂临床应用指南[J].中国医学前沿杂志(电子版), 2020, 12(5): 29-37.
Chinese Society of Gynecological Oncology, Chinese Medical Association. Guidelines for clinical application of PARP inhibitors in ovarian cancer[J]. Chin J Frontier Med (Electronic Edition), 2020, 12(5): 29-37
中国抗癌协会妇科肿瘤专业委员会. 中国卵巢上皮性癌维持治疗专家共识(2020)[J]. 中国实用妇科与产科杂志, 2020, 36(3): 234-238.
Gynecological Oncology Committee of China Anti Cancer Association. Expert consensus on maintenance treatment of epithelial ovarian cancer in China (2020)[J]. Chin J Pract Gynecol Obstetr, 020, 36(3): 234-238.
National Comprehensive Cancer Network (NCCN). Clinical Practice Guidelines in Oncology. Ovarian Cancer, Version 1. 2020.
RAY-COQUARD I, PAUTIER P, PIGNATA S, et al. Olaparib plus bevacizumab as first-line maintenance in ovarian cancer[J]. N Engl J Med, 2019, 381(25): 2416-2428.
GONZÁLEZ-MARTÍN A, POTHURI B, VERGOTE I, et al. Niraparib in patients with newly diagnosed advanced ovarian cancer[J]. N Engl J Med, 2019, 381(25): 2391-2402.
ALEXANDROV L B, NIK-ZAINAL S, WEDGE D C, et al. Signatures of mutational processes in human cancer[J]. Nature, 2013, 500(7463): 415.
NAIPAL K A, VERKAIK N S, AMEZIANE N, et al. Functional ex vivo assay to select homologous recombination–deficient breast tumors for PARP inhibitor treatment[J]. Clin Cancer Res, 2014, 20(18): 4816-4826.
DAVIES H, GLODZIK D, MORGANELLA S, et al. HRDetect is a predictor of BRCA1 and BRCA2 deficiency based on mutational signatures[J]. Nat Med, 2017, 23(4): 517.
《基于下一代测序技术的BRCA1/2基因检测指南(2019版)》编写组. 基于下一代测序技术的BRCA1/2基因检测指南(2019版)[J]. 中华病理学杂志, 2019, 48(9): 670-677.
Working Group of Guideline on Next-Generation Sequencing-Based BRCA1/2 Testing (2019). Guideline on next-generation sequencing-based BRCA1/2 testing (2019) [J]. Chin J Pathol, 2019, 48(9): 670-677.
BRCA数据解读中国专家共识编写组. BRCA数据解读中国专家共识[J]. 中华病理学杂志, 2017, 46(5): 293-297.
Working Group of Chinese experts consensus on the Interpretation of BRCA data. Chinese experts consensus on the Interpretation of BRCA data [J]. Chin J Pathol, 2017, 46(5): 293-297.
RICHARDS S, AZIZ N, BALE S, et al. Standards and guidelines forthe interpretation of sequence variants: a joint consensus recommendation of the American College of Medical Genetics and Genomics and the Association for Molecular Pathology[J]. Genet Med, 2015, 17(5): 405-424.
ENIGMA Consortium. ENIGMA BRCA1/2 gene variant classification criteria [ EB-OL]. Version 2.5.1 29 June 2017. https://enigmaconsortium.org/wp-content/uploads/2018/10/ENIGMA_Rules_2017-06-29-v2.5.1.pdf.
CHAKRAVARTY D, GAO J, PHILLIPS S M, et al. OncoKB: a precision oncology knowledge base [J]. JCO Precis Oncol, 201717.00011.
LI M M, DATTO M, DUNCAVAGE E J, et al. Standards and guidelines for the interpretation and reporting of sequence variants in cancer[J]. J Mol Diagn, 2017, 19(1): 4-23.
WATKINS J A, IRSHAD S, GRIGORIADIS A, et al. Genomic scars as biomarkers of homologous recombination deficiency and drug response in breast and ovarian cancers[J]. Breast Cancer Res, 2014, 16(3): 211.
ABKEVICH V, TIMMS K M, HENNESSY B T, et al. Patterns of genomic loss of heterozygosity predict homologous recombination repair defects in epithelial ovarian cancer[J]. Br J Cancer, 2012, 107(10): 1776-1782.
POPOVA T, MANIÉ E, RIEUNIER G, et al. Ploidy and large-scale genomic instability consistently identify basal-like breast carcinomas with BRCA1/2 inactivation[J]. Cancer Res, 2012, 72(21): 5454-5462.
BIRKBAK N J, WANG Z C, KIM J Y, et al. Telomeric allelic imbalance indicates defective DNA repair and sensitivity to DNA-damaging agents[J]. Cancer Discov, 2012, 2(4): 366-375.
STRONACH E A, PAUL J, TIMMS K M, et al. Biomarker assessment of HR deficiency, tumor BRCA1/2 mutations, and CCNE1 copy number in ovarian cancer: associations with clinical outcome following platinum monotherapy[J]. Mol Cancer Res, 2018, 16(7): 1103-1111.
FDA. Myriad myChoice CDx - P190014. [EB/OL].https://www.fda.gov/medical-devices/recently-approved-devices/myriad-mychoice-cdx-p190014.
FDA. FoundationFocus CDxBRCA LOH - P160018/S001 [EB/OL]. https://www.fda.gov/medical-devices/recently-approved-devices/foundationfocus-cdxbrca-loh-p160018s001.
国家药品监督管理局. 体外诊断试剂注册管理办法(国家食品药品监督管理总局令第5号)[EB/OL]. https://www.nmpa.gov.cn/xxgk/fgwj/bmgzh/20140730170001489.html.
国家药品监督管理局医疗器械技术审评中心. 肿瘤相关突变基因检测试剂(高通量测序法)性能评价通用注册技术审查指导原则(2019年第83号)[EB/OL]. https://www.nmpa.gov.cn/.
国家药品监督管理局医疗器械技术审评中心. 体外诊断试剂临床试验技术指导原则(2014年第16号)[EB/OL]. https://www.nmpa.gov.cn/xxgk/ggtg/qtggtg/20140911120001840.html.
FDA. In Vitro Companion Diagnostic Devices, document issued on August 6, 2014. [EB/OL]. https://www.fda.gov/regulatory-information/search-fda-guidance-documents/vitro-companion-diagnostic-devices.
FDA. Considerations for Design, Development, and Analytical Validation of Next Generation Sequencing (NGS) - Based In Vitro Diagnostics (IVDs) Intended to Aid in the Diagnosis of Suspected Germline Diseases, document issued on April 13, 2018[EB/OL]. https://www.fda.gov/regulatory-information/search-fda-guidance-documents/considerations-design-development-and-analytical-validation-next-generation-sequencing-ngs-based.
JENNINGS L, VAN DEERLIN V M, GULLEY M L, et al. Recommended principles and practices for validating clinical molecular pathology tests[J]. Arch Pathol Lab Med, 2009, 133(5): 743-755.
KHAN M U, BOWSHER R R, CAMERON M, et al. Recommendations for adaptation and validation of commercial kits for biomarker quantification in drug development[J]. Bioanalysis, 2015, 7(2): 229-242.
KONSTANTINOPOULOS P A, NORQUIST B, LACCHETTI C, et al. Germline and somatic tumor testing in epithelial ovarian cancer: ASCO guideline[J]. J Clin Oncol, 2020, 38 (11): 1222-1245.
TEW W P, LACCHETTI C, ELLIS A, et al. PARP inhibitors in the management of ovarian cancer: ASCO guideline[J]. J Clin Oncol, 2020: JCO2001924.
DALY M B, PILARSKI R, YURGELUN M B, et al.NCCN guidelines insights: genetic/familial high-risk assessment: breast, ovarian, and pancreatic, version 1.2020[J]. J Natl Compr Canc Netw, 2020, 18(4): 380-391.

 妇产科在线APP下载
妇产科在线APP下载












