引言
染色体非整倍体代表了一大类人类遗传缺陷,大约每300个活产婴儿中就发生1例。在活产婴儿中常见的染色体非整倍体有21三体、18三体、13三体和性染色体非整倍体。【1】 其中,21三体(唐氏综合征)的发病率为1/800。虽然18-三体和13-三体综合征在活产婴儿中的发生率比21三体低,分别为1/6000和1/100,000,但在流产胎儿中的比例却非常高。【2】性染色体异常在活产婴儿中的发生率大约为千分之一,常见的性染色体非整倍体有X单体(45,X或特纳氏综合征),XXY综合征(47,XXY或克氏综合征),XYY综合征(47,XYY)和Triple X综合征(47,XXX)。
虽然这些疾病由遗传物质改变引起,但检测手段仍依赖于间接的血清学筛查。【3】血清学筛查是一种综合评估方法,通过孕妇年龄、胎儿B超检查以及妊娠期间母体血液中相应蛋白和激素的变化水平,计算出胎儿患染色体疾病的风险值。 【4】没有任何一项血清学筛查指标是基于染色体上的基因表达产物或衍生物,因此它与遗传物质的改变有没有直接关系。【5,6】目前血清学筛查只针对21三体和18三体综合征,最有效的筛查灵敏度约为90%,假阳性率为2%至5%。而对其他染色体非整倍体的检测,只能采用侵入性的产前诊断方法(包括羊膜穿刺、绒毛膜取样、脐带穿刺术),对胎儿细胞染色体核型分析进行判断。然而,这些侵入性的诊断方法伴随着大约0.5%的流产风险。【7,8】随着母体外周血中胎儿游离DNA的发现【9】以及大规模平行测序技术的问世,【10-12】 使得胎儿染色体非整倍体的无创产前检测(NIPT)应运而生。【13-16】 这种方法首先实现了对21三体综合征的检测。【13-15】 最近几项临床研究结果表明,对21三体的检测灵敏度和特异度分别可达100%和99%。【17-22】对于18号和13号染色体来说,由于其变异系数(CV)明显大于21号染色体的变异系数,因此通过测序的方法检测18三体和13三体比21三体更加困难,【13,14】但根据文献记录,对GC含量进行校正之后,可以准确地检测到18三体和13三体【20,21,23】
材料与方法
研究设计和样品采集
从2009年3月到2011年6月,经过各院方审查委员会的批准,我们在湖南省妇幼保健院、湘雅医院、福建省妇幼保健院共招募了435位经传统筛查被判断为唐氏综合征高风险的孕妇参与本研究,每一位参与者都签署了知情同意书,并接受了进一步的侵入性产前诊断。在进行侵入性手术前,从每位参与者身上采集五毫升母体外周血,置于EDTA-K2 Vacutainer真空采血管中(美国新泽西州富兰克林湖市,Becton Dickinson公司),按照本文所描述的操作步骤离心,并将血浆运到北京贝瑞和康公司进行DNA提取和下游的测序分析。表S1总结了参与者的统计资料, 包括其指征分类:217个样本来自血清学筛查(49.89%),84个样本来自高龄产妇(19.31%),67个样本来自超声波异常,4个样本来自非整倍体妊娠史,还有64个样本是来自具有多重指征的患者。除1例样本为孕11周外,其余样本的孕周范围为15周到39周。其中174位参与者为15+0到20+6孕周,260位为21+0到39+6孕周。胎儿细胞的染色体核型分析由湖南省妇幼保健院、湘雅医院和福建省妇幼保健院的相应科室进行,核型分析结果见表S2。母血浆DNA测序及结果分析由北京贝瑞和康公司独立完成,在分析完成之前,所有样本的核型信息都保持盲测状态。
为本研究免费完成测序分析的贝瑞和康公司,是一家提供无创产前检测和基因测序服务的中国服务商。本文作者在整个课题中进行了样本采集、侵入性手术和染色体核型分析、结果揭盲等工作,并参与了最后的数据分析。
母体血浆DNA处理和测序
采集母体血样后的8个小时内,在4oC条件下以1600g转速离心10分钟。将血浆转移至微量离心管中,在4oC条件下以16000g转速再离心10分钟,以去除剩余的细胞,然后转移到新的离心管中,并置于-80oC保存。每个样本取1ml血浆进行DNA抽提,使用试剂为Qiagen公司(德国希尔登市)生产的QIAmp循环核苷酸试剂盒。将得到的血浆DNA作为模板,进行文库构建,所使用的方法为改良的染色质免疫共沉淀测序(ChIP Seq)法。13 ,使用6bp的寡核苷酸标签分别对8-12个血浆DNA文库进行标记(indexed),并使用卡帕生物系统公司(美国马里兰州沃本市)生产的KAPA SYBR快速qPCR试剂盒进行定量,混合后加入Illumina公司HiSeq2000测序系统v2流动槽的其中一个通道(lane)中。按照Illumina公司的产品说明书,使用43bp单端测序法生成分子簇并进行测序。
数据分析
根据标签,每一个样本的测序结果都独立生成一个对应的数据文件(.bin的文件),并比对到已知的人类基因组序列。我们测试了几种比对软件,包括BWA、【24】 Bowtie【25】和SOAP2。【26】 发现这三种比对算法的结果大致相当。为与先前报道一致,【27】 我们采用了通过SOAP2比对得到的结果。有23个样本没有达到质量控制标准,其中12个样本测序失败,另外11个样本为染色体核型分析失败(图S1)。对于剩余的412个样本,我们统计了比对到每个染色体的序列,并计算了其GC含量。并从一组正常的46,XX胎儿的染色体核型独立样本集产生由50个女性二倍体样本组成的参照集。
对于每个样本的每条染色体,按照以下公式生成以百分比为单位的归一化染色体表达(NCR)值:NCR = 唯一比对到目标染色体的序列数/唯一比对所有常染色体的序列总数。 【18】 对于除了50个参照样本中的14号染色体和Y染色体之外的所有染色体,我们将其NCR值对GC含量作图,并用简单线性回归计算斜率。通过斜率对412个通过了质量控制的样本进一步进行归一化处理:NCRgc =NCR - (GC - GCaverage_ref)/斜率ref,此处NCRgc为经GC含量校正的NCR值,NCR为未经校正的数值,GC为测试样本的染色体GC含量,GCaverage_ref与斜率ref分别为参照样本的平均GC含量以及斜率。我们计算了参照数据集的NCRgc均值(μref)和标准偏差(δref)。j样本的i号染色体(i可以是24种染色体中除14号染色体和Y染色体外的任何一种)的z值由以下公式计算得出:zij =(xij - μref)/ δref,此处xij为j样本的i号染色体的NCRgc值。对于14号染色体和Y染色体,计算z值时没有针对GC含量做校正。对于任何一个男性胎儿j样本,胎儿DNA的百分比基于以下公式估算得出:2 * (1 - xj),此处xj为男性胎儿j样本的X染色体的NCRgc与女性胎儿参照样本的X染色体的平均NCRgc之间的比例。【28】
结果
母体血浆DNA测序
本研究共采集了435份外周血样本,提取血浆DNA并进行测序。试验中,我们对每个样本都进行了标签标记,然后将8到12个样本混合,装入八通道流动槽的其中一个通道中。为测试通道与通道之间以及每次测序运行之间的差异,我们在每个通道中都加入了两个对照样本:一个为二倍体样本(D21),另一个为21三体样本(T21)。测序结果共产生了543个数据项,其中108个数据项由两个对照样本产生,各含54个数据点,且各来自于同一个DNA文库。对于每个样本,平均收集到了830万条长度为36bp的DNA序列信息,并将得到的序列信息比对到已知的人类基因组上(hg19)。对于每个染色体,我们通过将唯一比对到目标染色体上的序列数除以唯一比对到所有常染色体上的序列总数来计算NCR值。【18】 通过分析对照样本,我们注意到,不同批次的测序所产生的数据点之间,以及在单次测序的不同通道之间,NCR值的波动范围很大。为寻找波动的原因,我们将唯一比对到21号染色体的序列的NCR值对其GC含量作图,结果观察到在NCR值和GC含量之间存在着一种线性关系(图1A)。进一步的观察发现,除了14号染色体之外,类似的线性关系也存在于其他种染色体之中,但斜率和截距有所不同。为了消除这种GC含量带来的干扰,我们根据该线性关系用GC含量校正了NCR值(见方法章节)。进行GC含量校正之后,除了12号染色体、21号染色体和Y染色体之外,其他染色体的变异系数都有显著降低(图1B),尤其是3号、4号、5号、6号、13号、16号、17号、18号、19号、20号、22号染色体及X染色体的变异系数降低了57%到82%(表S3)。
胎儿21三体的无创检测
在去掉对照样本的重复数据点之后,我们分析了412个非重复性数据点,它们代表着通过了质量控制标准的412个样本(图S1)。通过将50个女性样本组成的数据集作为参照,进行上述的GC含量校正后,我们计算出了每种常染色体的z值(z-score)。【13】当z-score的切割值设定为3时,可以识别出40个T21综合征样本(图2B),这些样本的数据点在NCR对GC含量所做的散点图中与D21样本是清楚分开的(图2A)。通过测序识别出的40个T21样本,与通过核型分析识别出的40个T21样本完全匹配,检测灵敏度和特异度都达到了100%(表1)。同时在这40个T21样本中,包括1例嵌合体(47,XY,+21[26]/46,XY[24])及2例罗伯逊易位,这一结果证实了先前的报告,即由罗伯逊易位引起的21三体可以用测序的方法检测到。【29】T21样本的最低z-score值为3.89,而D21样本的最高z-score为2.24。
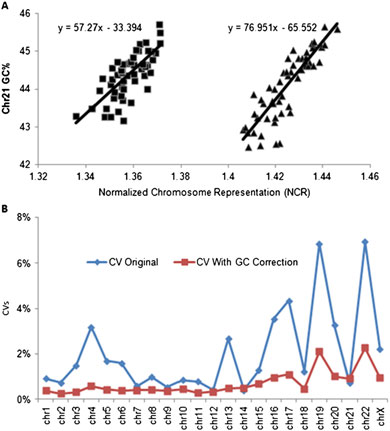
图1(A)归一化的染色体表达(NCR)值与GC含量之间的线性关系。对于重复的对照样本,在NCR值与唯一比对到21号染色体上的序列标签的GC含量之间存在着线性关系。每个方块(D21样本)或三角形(T21样本)各代表一个数据点。(B)GC校正降低了检测的变异系数(CV)。除了12号、21号染色体和Y染色体之外,GC校正降低了所有24条染色体的检测变异系数。
胎儿18三体的无创检测
用同样的方法对18号染色体进行检测,我们发现18号染色体的检测变异系数(0.47%)比21号染色体低(0.92%)。当21号染色体z-score的切割值为3时,母血浆中胎儿游离DNA含量为5.52%,而当胎儿DNA处于这个水平时,计算出的18三体样本的z-score为5.91。于是我们将18三体的z-score切割值设为5.91(表S3)。
测序数据显示,有14个样本的18号染色体z-score值高于5.91(图2D),经验证,这14个样本与染色体核型分析结果完全一致,其中还包括1例嵌合体样本(46,XX[96]/47,XX,+18[3]),对18号染色体的检测灵敏度达到100%(14/14),特异度也达到100%(表1)。在NCR对GC含量的散点图上,T18和非T18样本的数据点也同样清晰地相互分开(图2C)。T18样本的z-score范围为6.46到34.33,而非T18样本的最高z-score为3.72。
胎儿13三体的无创检测
本研究中,经染色体核型分析确定了4个T13样本,包括一对双胞胎样本(双胎中的一个是13三体)。按照18三体的分析方法,当母血浆中胎儿DNA含量为5.52%时,我们计算得到13三体的z-score 切割值为5.72(表S2)。当以5.72为z-score的切割值时,可以判断出5个T13的样本,其中4个样本与染色体核型分析结果正确匹配,达到了100%的检测灵敏度以及99.75%(407/408)的特异度(表1)。T13样本的z-score范围为7.17到19.54,其中双胞胎样本的z-score最低(图2E、2F)。对于假阳性的T13样本,经核型证实是一个45,X样本,其z-score为5.81。

图2(A)胎儿21三体的无创检测。将归一化的染色体表达(NCR)值对唯一比对到21号染色体的序列的GC含量作图。三角形代表T21样本,菱形代表罗伯逊易位,方块代表嵌合体,圆圈代表D21样本。(B)D21(左)和T21(右)样本的z-score。虚线代表z-score的切割值为3。(C)胎儿18三体的无创检测。三角形代表T18样本,方块代表嵌合体,而圆圈代表D18样本。(D)D18(左)和T18(右)样本的z-score。虚线代表z-score的临界值为5.91。(E)胎儿13三体的无创检测。三角形代表T13样本,菱形代表双胞胎样本,而圆圈代表D13样本。(F)D13(左)和T13(右)样本的z-score。虚线代表z-score的切割值为5.72。(G)胎儿9三体的无创检测。三角形代表T9样本,而圆圈代表D9样本。(H)D9(左)和T9(右)样本的z-score。虚线代表z-score的切割值为7.45。
其他常染色体染色体非整倍体的无创检测
上述原则同样适用于其他常染色体。在本研究中,通过上述分析方法发现了一例9-三体样本。将9号染色体的z-score切割值设为7.45时(基于母血浆中胎儿DNA含量为5.52%,见表S3),准确地判断出了9-三体样本,且未产生假阳性样本(图2G)。T9样本的z-score为11.11,而412例中的其余样本的z-score范围为-2.66到2.45(图2H)。
表1 本研究中检测到的非整倍体
综合征 | 检测灵敏度 | 检测特异度 | PPV | NPV |
21三体 | 100% (40/40) | 100% (372/372) | 100% (40/40) | 100% (372/372) |
18三体 | 100% (14/14) | 100% (398/398) | 100% (14/14) | 100% (398/398) |
13三体a | 100% (4/4) | 99.75% (407/408) | 80% (4/5) | 100% (407/407) |
9三体 | 100% (1/1) | 100% (411/411) | 100% (1/1) | 100% (411/411) |
45, Xb | 100% (5/5) | 99.75% (406/407) | 83.3% (5/6) | 100% (405/405) |
XXX | 100% (1/1) | 100% (411/411) | 100% (1/1) | 100% (411/411) |
XXY | 100% (1/1) | 100% (411/411) | 100% (1/1) | 100% (411/411) |
XYY | 100% (1/1) | 100% (411/411) | 100% (1/1) | 100% (411/411) |
综合检测 | 100% (67/67) | 99.71% (344/345) | 98.53% (67/68) | 100% (343/343) |
PPV:阳性预测值;NPV:阴性预测值。
a 包括一对双胞胎样本。
b 不包括三个嵌合体45,X样本。
胎儿性染色体非整倍体的无创检测
我们用同样的方法计算了性染色体的NCR值和z-score。当Y染色体的z-score切割值设为3时,可以准确地区分男性样本和女性样本,灵敏度和特异度均达到100%。当X染色体的z-score切割值设为-2.91时(基于母血浆中胎儿DNA含量为5.52%的水平,见表S3),能准确识别出211个女性样本,但将2个男性样本错判为女性,特异度为99.51%。考虑到性染色体的拷贝数与母体血浆中的胎儿DNA含量成正比,我们认为男性样本X染色体和Y染色体的z-score值应互为线性关系,并存在检测误差和随机取样误差。通过将X染色体的z-score对Y染色体的z-score作图后发现,男性样本性染色体的z-score确实是呈正相关的(图3)。对于女性样本,我们预测性染色体的z-score会围绕原点(0,0)分布:对于X染色体,z-score应在-2.91到+2.91的范围内;对于Y染色体,z-score应小于3。事实证明,结果与我们所预测的一样(图3)。而在这两个数据范围之外的,就有可能是性染色体非整倍体样本。根据这个原则,我们识别出7个可能患有特纳综合征(45,X)的样本,这些样本的X染色体的z-score低于-2.91,Y染色体的z-score小于3。经核型验证,在这7个样本中,5个样本与染色体核型分析完全一致;1个为特纳综合征嵌合体样本:45,X[88]/46,XY[13];还有一个为假阳性样本,这例假阳性样本有几个染色体的z-score值都不正常。此外,通过测序还检测出一个XXY样本,一个XYY样本,以及一个XXX样本(图3),这些结果都与染色体核型分析结果相一致。此外,有2个特纳综合征嵌合体样本没能通过测序检测出来:45,X[9]/46,XX[41]和45,X[48]/46,XX[15]。除嵌合体样本外,对性染色体非整倍体而言,上述结果达到了100%(8/8)的灵敏度和99.75%(403/404)的特异度(表1)。

图3(A)胎儿性染色体非整倍体的无创检测。将X染色体的z-score(X轴)对Y染色体的z值(Y轴)作图,以显示男性样本(蓝色男性符号)、女性样本(红色女性符号)、XYY样本(红色实点)、XXY样本(绿色菱形)、45,X样本(绿色三角形和绿色方块)及XXX样本(黑色椭圆形)。绿色方块为45,X嵌合体样本。(B)图3A的放大版。
讨论
通过母体血浆DNA测序进行无创胎儿染色体非整倍体产前检测,已有多次报道。【13–23,27–29】 但这些研究大多仅局限于三种常染色体。Chen EZ等人首先通过回归性的实验数据而集中推导出局部GC含量校正算法,实现了对13三体和18三体的有效检测,【27】——尽管他在37例T18样本中,有3例漏检。 Palomaki等人的后续研究也进一步证实了GC含量校正算法对13三体和18三体检测的有效性。【20】
本研究中,我们希望通过母体血浆DNA测序技术实现对胎儿所有染色体非整倍体的检测。通过对412份母体血浆游离DNA测序,共检测到了67个非整倍体样本,覆盖了6种染色体(表1)。检测中出现2例假阳性,其中1例为T13假阳性,但却是一个真实的45,X阳性样本。将这6种染色体作为整体考虑,可以得到结论:本研究检测到了全部67个非整倍体样本,有1例假阳性,达到100%(67/67)的检测灵敏度以及99.71%(344/345)的特异度。试验结果表明,基于GC含量校正后的无创DNA产前检测技术,可以实现对胎儿所有染色体非整倍体的检测。
本实验同时证明,通过GC含量校正可以大幅度提高检测的灵敏度。在数据分析过程中,我们最初采用了Chiu等人开发的标准z-score算法,【13】 这种算法没有进行GC含量校正,结果发现,虽然能将40个T21样本全部检出,但同时产生2个假阳性;同时可以检测出13例18三体和3例13三体,虽无假阳性,但却各存在一例漏检。正如先前的报道所说,【14,27】 无创产前检测(NIPT)对于每一种染色体的变异系数的要求都很高。本文中所使用的GC含量校正法可以大幅降低绝大多数染色体的变异系数,从而提高检测的灵敏度。当然,检测灵敏度也依赖于胎儿DNA在母血中的百分比含量。在分析201个男性胎儿样本DNA在母血浆中含量时(图S2)发现,有199个样本的胎儿DNA百分比含量超过5.52%。 本试验中,用这一数值来设定z-score切割值,这就意味着按照本文中提出的z-score切割值,99%的样本血浆DNA含量是足以检测到21三体、18三体、13三体、9三体和性染色体倍性异常的。换言之,这些染色体的检测灵敏度应该在99%以上,与当前检测灵敏度为90%的筛选方法相比,这是一个突破。虽然缺少早孕期相关的数据支持,但由于胎儿DNA在母血浆中的含量从早孕期的后期到中孕期几乎是恒定的(20,30),因此,我们认为,以这样的灵敏度和特异度,无创产前检测技术应适用于早孕期后期的样本。
本研究证明了在临床上应用无创DNA产前检测的可行性。我们采用了12-plex 的测序芯片,对多达12个样品进行标签区分(index)后混合到Illumina测序系统V2流动槽的一个测序通道中,不仅大大降低了测序成本,同时也将通量提高到每台HiSeq2000(Illumina公司)测序仪年处理30000个样本的水平。在实践中,测序试剂和仪器都处于不断升级的进程中,但上述GC含量校正算法,始终可以应用于测序结果的分析,只是每次测序必须从新计算参照集和GC含量校正参数。可以预见,随着测序通量不断提高,成本持续下降,无创DNA产前检测的成本肯定会变得更低。
结论
研究发现,所有染色体的基因组与其GC含量之间都符合线性关系。利用这种关系,我们可以通过一次测序,准确判断出包含21三体、18三体、13三体、9三体、X单体在内的胎儿染色体非整倍体样本,达到100%的检测灵敏度和99.71%的检测特异度。我们相信,基于高通量测序的无创产前检测技术未来很有希望应用于临床。
致谢
感谢贝瑞和康公司的团队进行了测序和分析工作。我们十分感谢所有参与本研究的孕妇。感谢医院的工作人员协助样本采集以及进行染色体核型分析。
对于这个话题我们已经知道些什么?
-- 随着母体外周血中存在着胎儿游离DNA片段的发现以及大规模平行测序技术的问世,通过无创产前检测(NIPT)排查胎儿染色体非整倍体已经成为可能。
本研究增加了什么新知识?
-- 本研究发现,所有染色体的基因组表达与其GC含量之间都符合线性关系。利用这种关系,我们分析了435个临床样本中24种染色体各自的拷贝数,结果显示,本文所使用的方法对胎儿染色体非整倍体的检测达到了100%的检测灵敏度和99.71%的特异度。本试验采用了可同时测试12个样本的芯片(12-plex protocol),这证明,基于大规模平行测序技术的无创产前检测法在通量上有进一步提升的空间。
参考文献
1 Driscoll DA, Gross S. Clinical practice.Prenatal screening for aneuploidy.N Engl J Med 2009;360:2556–62.
2 Griffiths A, Miller J, Suzuki D, et al.Chromosome mutation II:Changes in chromosome number.An Introduction to Genetic Analysis (7th edn).New York:W. H. Freeman, 2000.
3 Wald NJ, Watt HC, Hackshaw AK.Integrated screening for Down’s syndrome on the basis of tests performed during the first and second trimesters.N Engl J Med 1999;341:461–7.
4 Malone FD, Canick JA, Ball RH, et al.First-trimester or second-trimester screening, or both, for Down’s syndrome.N Engl J Med 2005;353:2001–11.
5 Palomaki GE, Haddow JE, Knight GJ, et al.Risk-based prenatal screening for trisomy 18 using alpha-fetoprotein, unconjugated oestriol and human chorionic gonadotropin.Prenat Diagn 1995;15:713–23.
6 Crandall BF, Hanson FW, Keener S, et al.Maternal serum screening for alpha-fetoprotein, unconjugated estriol, and human chorionic gonadotropin between 11 and 15 weeks of pregnancy to detect fetal chromosome abnormalities.Am J Obstet Gynecol 1993;168:1864–7; discussion 1867-9.
7 Odibo AO, Dicke JM, Gray DL, et al.Evaluating the rate and risk factors for fetal loss after chorionic villus sampling.Obstet Gynecol 2008;112:813–9.
8 Odibo AO, Gray DL, Dicke JM, et al.Revisiting the fetal loss rate after second-trimester genetic amniocentesis: a single center’s 16-year experience.Obstet Gynecol 2008;111:589–95.
9 Lo YM, Corbetta N, Chamberlain PF, et al.Presence of fetal DNA in maternal plasma and serum.Lancet 1997;350:485–7.
10 Bentley DR, Balasubramanian S, Swerdlow HP, et al.Accurate whole human genome sequencing using reversible terminator chemistry.Nature 2008;456:53–9.
11 Mardis ER.A decade’s perspective on DNA sequencing technology.Nature 2011;470:198–203.
12 Metzker ML.Sequencing technologies – the next generation.Nat Rev Genet 2010;11:31–46.
13 Chiu RW, Chan KC, Gao Y, et al.Noninvasive prenatal diagnosis of fetal chromosomal aneuploidy by massively parallel genomic sequencing of DNA in maternal plasma.Proc Natl Acad Sci USA 2008;105:20458–63.
14 Fan HC, Blumenfeld YJ, Chitkara U, et al.Noninvasive diagnosis of fetal aneuploidy by shotgun sequencing DNA from maternal blood.Proc Natl Acad Sci USA 2008;105:16266–71.
15 Chiu RW, Sun H, Akolekar R, et al.Maternal plasma DNA analysis with massively parallel sequencing by ligation for noninvasive prenatal diagnosis of trisomy 21.Clin Chem 2010;56:459–63.
16 Lo YM.The quest for accurate measurement of fetal DNA in maternal plasma.Clin Chem 2011;57:522–3.
17 Chiu RW, Akolekar R, Zheng YW, et al.Non-invasive prenatal assessment of trisomy 21 by multiplexed maternal plasma DNA sequencing: large scale validity study.BMJ 2011;342:c7401.
18 Ehrich M, Deciu C, Zwiefelhofer T, et al.Noninvasive detection of fetal trisomy 21 by sequencing of DNA in maternal blood: a study in a clinical setting.Am J Obstet Gynecol 2011;204:205 e201–11.
19 Sehnert AJ, Rhees B, Comstock D, et al.Optimal detection of fetal chromosomal abnormalities by massively parallel DNA sequencing of cell-free fetal DNA from maternal blood.Clin Chem 2011;57:1042–9.
20 Palomaki GE, Deciu C, Kloza EM, et al.DNA sequencing of maternal plasma reliably identifies trisomy 18 and trisomy 13 as well as Down syndrome: an international collaborative study.Genet Med 2012;14:296–305.
21 Palomaki GE, Kloza EM, Lambert-Messerlian GM, et al.DNA sequencing of maternal plasma to detect Down syndrome: an international clinical validation study.Genet Med 2011;13:913–20.
22 Sparks AB, Wang ET, Struble CA, et al.Selective analysis of cell-free DNA in maternal blood for evaluation of fetal trisomy.Prenat Diagn 2012;32:3–9.
23 Bianchi DW, Platt LD, Goldberg JD, et al.Genome-wide fetal aneuploidy detection by maternal plasma DNA sequencing.Obstet Gynecol 2012;119:890–901.
24 Li H, Durbin R. Fast and accurate short read alignment with Burrows– Wheeler transform.Bioinformatics 2009;25:1754–60.
25 Langmead B, Trapnell C, Pop M, et al.Ultrafast and memory-efficient alignment of short DNA sequences to the human genome.Genome Biol 2009;10:R25.
26 LiR,YuC,LiY, et al.SOAP2: an improved ultrafast tool for short read alignment.Bioinformatics 2009;25:1966–7.
27 Chen EZ, Chiu RW, Sun H, et al.Noninvasive prenatal diagnosis of fetal trisomy 18 and trisomy 13 by maternal plasma DNA sequencing.PLoS One 2011;6:e21791.
28 Fan HC, Quake SR.Sensitivity of noninvasive prenatal detection of fetal aneuploidy from maternal plasma using shotgun sequencing is limited only by counting statistics.PLoS One 2010;5:e10439.
29 Lun FM, Jin YY, Sun H, et al.Noninvasive prenatal diagnosis of a case of Down syndrome due to Robertsonian translocation by massively parallel sequencing of maternal plasma DNA.Clin Chem2011;57:917–9.
 妇产科在线APP下载
妇产科在线APP下载













