作者:
Xiaolin Shi a, Zhitao Zhang a, David S. Cram b,*, Caixia Liu a,**
a中国医科大学附属盛京医院妇产科,辽宁沈阳,中国
b北京贝瑞和康生物技术有限公司,北京,中国
*共同通讯作者,**通讯作者
摘要:
背景: 非侵入性产前检测(NIPT)运用大 规 模 平 行 测 序 技 术(MPS)检测妊娠中期外周血胎儿游离DNA(cffDNA),现已作为常见胎儿染色体疾病的一线检测技术加以运用。若在妊娠早期知晓NIPT检测结果,对于高危妊娠人群的临床管理更为有利。本研究旨在验证NIPT用于妊娠早期胎儿染色体疾病检测的可行性。
方法:构建用于MPS的血浆DNA文库,以参考基因组为参照进行染色体片段的标准化,基于z值大小确认样本是否为染色体非整倍体(正常值为-3
结论:NIPT用于妊娠早期常见胎儿染色体非整倍体的检测,高度可靠且准确,但由于少数孕妇cffDNA含量过低,因而临床应用仍需谨慎。
关键词:妊娠早期、胎儿染色体非整倍体、非侵入性产前检测、胎儿游离DNA
背景
非侵入性产前检测(Non-invasive Prenatal Diagnosis Testing,NIPT)运用大 规 模 平 行 测 序 技 术(Massively Parallel Sequencing,MPS)检测外周血胎儿游离DNA(cell free fetal DNA,cffDNA),现已作为常见胎儿染色体疾病(如T21、T18、T13和性染色体非整倍体)的一线检测方法[1,2]加以运用。前瞻性临床研究表明[3–6],NIPT对T21、T18和T13具有很高的检测灵敏度和特异性,假阳性率和假阴性率低于0.1%。NIPT现已作为高危和低危孕妇妊娠中期的常规检测,偶用于早至10孕周的妊娠早期检测[2,7]。
cffDNA主要来源于胎盘细胞[8,9],其含量高低影响NIPT的检测精度。绝大多数情况下,NIPT可对cffDNA检测阈值(约4%)以上的样本实现精确检测。妊娠中期的cffDNA平均含量为10–21%,并以每周约1%速度递增[10,11]。孕期体重增加[10,12,13]、孕龄增加[10,12]、多胎妊娠[14,15]和先兆子痫[11,16] 是导致cffDNA含量较高的主要因素。此外,T21胎儿[17]、体能锻炼[18]、多种胎儿特征[13]和不良妊娠结局[11]等次要因素也参与调节cffDNA含量。不过上述所有因素均不能用于预测cffDNA含量,揭示存在其它因素参与胎儿及母亲的最终外周血DNA比例调节[11]。
NIPT对妊娠早期进行检测,利于高危孕妇且能缓解孕妇等待妊娠中期检测结果的焦虑。本研究旨在评估NIPT用于妊娠早期的可行性。共有182名妊娠早期(8-12孕周)和妊娠中期(15-18孕周)的高危孕妇参与本次研究。研究结果显示,NIPT对于妊娠早期低水平的cffDNA仍具有较好的检测可靠性和精确性。不过与现有理论相悖的是,本研究首次揭示单个患者的cffDNA含量也可能出现较大波动。从妊娠早期至中期,cffDNA含量既可能升高,又可能降低,或维持不变。
方法
试验设计
本研究已取得中国医科大学附属盛京医院伦理委员会批准。共209名高危孕妇参加本研究,包括高龄和(或)有流产史的孕妇。所有接受NIPT检测的孕妇均需签署知情同意书,并分别于妊娠早期(8-11+6孕周)和中期(15-18孕周)采集外周血样本。NIPT阳性孕妇需在15-20孕周接受羊膜腔穿刺术和核型分析。
样本采集和处理
使用Streck真空采血管抽取10ml外周血,并于48h内进行血浆分离:将Streck真空采血管以1600×g 离心10min。随后,转移血浆层至微量离心管,并以16000×g再次离心10min。小心转移血浆样品至另一支微量离心管。纯化的血浆样品-20℃保存。
NIPT
利用QIAamp血液DNA提取试剂盒(Qiagen公司)提取每份样本的血浆DNA。带有序列标签的接头引物用于构建血浆DNA文库。利用Illumina Hiseq2000平台进行MPS。根据已发表文献方法进行数据分析[3,19]。通过z值(正常值:-3<< span="">z<3< span="">)确定胎儿的染色体是否存在染色体非整倍体。如前所述,根据Y染色体序列片段所占比例,计算所有男性胎儿的游离DNA含量[20]。
结果
NIPT检测妊娠早期及中期样本
209名高危孕妇参加本研究并进行妊娠早期NIPT检测。NIPT检出9例染色体非整倍体,包括4例T21、4例T13和1例45,X(表1)。其中3例T21和3例T13后续流产。由于未采集到任何流产组织,因而未能确认相应的NIPT检测结果。在剩余的203名孕妇中,21名后续失访。因而总共182名孕妇进行妊娠中期NIPT检测,检测出1例T21、1例T13和1例45,X,而其余孕妇NIPT检测结果正常。
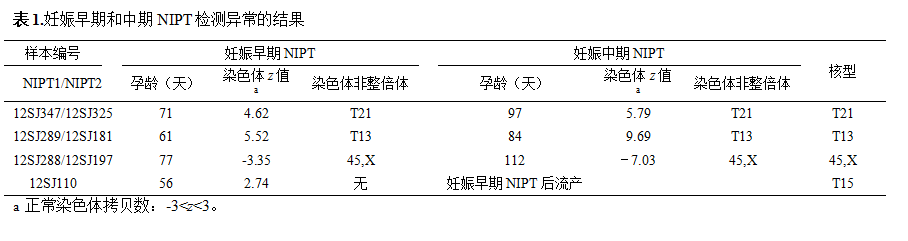
NIPT妊娠中期的检测结果与妊娠早期一致(表1),且经后续的羊膜腔穿刺术和核型分析确认。故而该3名孕妇后续选择终止妊娠。在妊娠早期NIPT检测结果正常的179名孕妇中,178名妊娠中期NIPT结果也正常,提示NIPT对于T21、T13和45,X的检测灵敏度和特异性接近100%。剩余1名孕妇在妊娠中期采样前已流产。随后对流产组织进行CNV-seq分析,确认为T15(图1)。重新对该患者的NIPT结果进行分析,发现15号染色体的z值在正常范围内(表1),为NIPT假阴性样本。
妊娠早期和中期的cffDNA含量分析
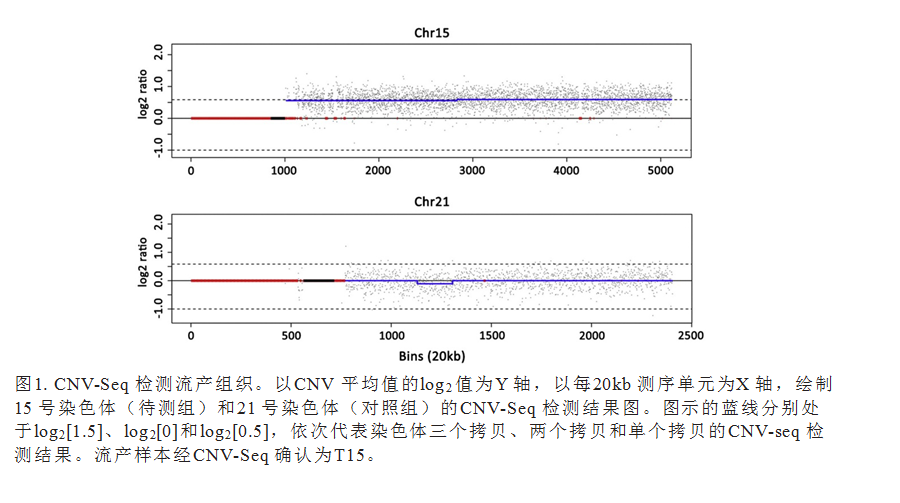
在重复进行NIPT检测的181名孕妇中,96名(53%)怀有男性胎儿,分析这些孕妇在妊娠早期和中期的cffDNA含量变化趋势。发现妊娠早期cffDNA的平均含量为7.6±4.18%,范围区间为2.56–21.03%(图2A)。随孕龄增加,妊娠第51–84天cffDNA含量轻微上升(R2=0.109,P<0.001< span="">)。值得注意的是,15名(15.6%)孕妇妊娠早期的cffDNA含量低于NIPT检测阈值4% [3,19],且孕龄随机分布。妊娠中期cffDNA平均含量为10.47±4.70%,范围区间为3.93–31.45%。95名(99%)孕妇的cffDNA含量高于4%(图2B)。随孕龄增加,妊娠中期cffDNA含量稍有上升(R2=0.005),但无统计学差异(P=0.48),怀疑由于样本采集时间过于集中(第105–120天)导致。综上所述,妊娠中期cffDNA平均含量高于妊娠早期,两者差异未显示统计学意义(P>0.05)。
cffDNA含量在妊娠早期和中期的差异比较
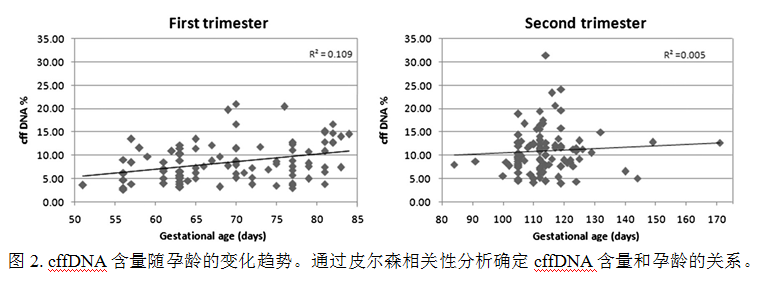
在NIPT分析96名怀有男性胎儿孕妇在妊娠早期和中期cffDNA含量的基础上,本研究进一步分析单一孕妇cffDNA含量的动态变化趋势(图3)。以1% cffDNA含量变动为参考,57名孕妇(59%)cffDNA含量上升,16名孕妇(17%)cffDNA含量不变,23名孕妇(24%)cffDNA含量下降。在cffDNA含量下降的23名孕妇中,78%降幅超过3%。有趣的是,15名妊娠早期cffDNA含量低于4%的孕妇在妊娠中期cffDNA含量明显升高,远高于4%这一阈值。
讨论
本研究共纳入209名高危孕妇评估NIPT在妊娠早期连续4周内的检测性能。受检孕妇在妊娠早期和妊娠中期均经NIPT检测,阳性孕妇后续进行核型分析确认。结果发现,NIPT在妊娠早期仍可取得与妊娠中期相近的检测精确度和可靠性[3–6]。本研究共检出3例T21,并经妊娠中期NIPT和核型分析确认。其余孕妇在妊娠早期和中期的NIPT检测结果均为阴性,显示NIPT具有很高的检测特异性。若不考虑样本量较小的因素,本研究显示NIPT对T21、T13和45,X的检测灵敏度为100%。此外,妊娠早期NIPT检测结束后自然流产的6名孕妇为T21或T13胎儿,进一步支持NIPT对于T21和T13具有较高的检测灵敏度。不过仍有1例T15在妊娠早期NIPT未检出,怀疑与该孕妇cffDNA含量过低有关。换言之,NIPT检测可能对T15无效,从而造成漏检[3,19,22]。
96名怀有男性胎儿的孕妇cffDNA平均含量在妊娠早期(第8–12孕周)为7.6%,妊娠中期为10.47%。后者与其它研究10–21%的妊娠中期cffDNA含量一致[10,11]。随着孕龄增加,cffDNA含量分布相对随机,揭示纳入本研究的中国孕妇虽然具有相近的体重和人口统计学特征,但人群异质性仍很明显。其中15%样本的cffDNA含量低于T21的检测阈值4%[3,19],3%样本cffDNA含量低于T18和T13的检测阈值3%[3,19]。若假设男性胎儿和女性胎儿的cffDNA含量相近,则严格意义上讲,NIPT依次不能检出妊娠早期(8-12孕周)15%的T21样本和3%的T18/T13样本。
通过逐个对比96名孕妇的妊娠早期和妊娠中期cffDNA含量变化,我们发现了此前尚未报道的cffDNA含量变化趋势。虽然59%孕妇的cffDNA含量明显升高,与现有理论一致[10,11],但仍有17%孕妇的cffDNA含量无明显变化,甚至24%孕妇的cffDNA含量下降。在cffDNA含量下降的23名孕妇中,cffDNA含量经过29天和64天分别下降9.91%和13.88%。我们推测其中2名孕妇的cffDNA含量降低与双胎消失综合征有关[23],而其余则可能与孕期体重剧增而胎盘重量仅平稳增加有关。不过本研究并未对孕期体重进行测量,因而上述推断需进一步数据的支持。我们需要更大样本量的前瞻性临床研究,按照严格的实验设计,多时间点取样,同时记录包括孕期体重增值、胎盘重量增值、胎盘功能和母胎接触面血液流速在内的多种参数,进而才有可能发现影响妊娠初期cffDNA含量变化的关键因素[11]。
本研究发现不同孕妇妊娠早期的cffDNA含量差异显著。约15%孕妇的cffDNA含量低于4%。使得在妊娠早期应用NIPT颇具挑战。初步解决方案是仅当妊娠早期cffDNA含量大于4%时才出具NIPT检测报告,而对于cffDNA含量低于4%的孕妇则被要求在妊娠中期再次接受NIPT检测。另一种更为合理的做法是提高NIPT对于低水平cffDNA的检测敏感度。近期出现的两种方法有望将cffDNA的检测水平降低至4%以下。一是对血浆DNA进行更深度测序,据报道该法已经能够在cffDNA含量为3%时检出T21 [24]。不过相应的检测成本会大大增加。二是利用了胎儿与母体的血浆DNA分子在整体片段大小方面的差异性[25],借助基于片段大小差异的MPS法,确定cffDNA低水平下的胎儿染色体非整倍体[26]。随着对于低水平cffDNA含量更高灵敏度检测方法的出现,NIPT将会应用于大多数孕妇的妊娠早期检测。
综上,本研究揭示NIPT可用于妊娠早期的染色体非整倍体检测,但低水平cffDNA检测问题尚待解决,临床应用仍需谨慎。不过毫无疑问,随着未来技术的发展,妊娠早期NIPT有望取得与妊娠中期相近的检测灵敏度与精确度,大大缩短诊断结果的等待时间,缓解孕妇及家属的心理压力。
参考文献
[1] Tsui NB, Lo YM. Recent advances in the analysis of fetal nucleic acids in maternal plasma. Curr Opin Hematol 2012;19:462–8.
[2] Bianchi DW, Wilkins-Haug L. Integration of noninvasive DNA testing for aneuploidy into prenatal care: what has happened since the rubber met the road? Clin Chem 2014;60:78–87.
[3] Song Y, Liu C, Qi H, Zhang Y, Bian X, Liu J. Non invasive prenatal testing of fetal an- euploidies by massively parallel sequencing in a prospective Chinese population. Prenat Diagn 2013;33:700–6.
[4] Bianchi DW, Parker RL, Wentworth J, et al. DNA sequencing versus standard prenatal aneuploidy screening. New Engl J Med 2014;370:799–808.
[5] Lau TK, Cheung SW, Lo PS, et al. Non-invasive prenatal testing for fetal chromosomal abnormalities by low-coverage whole-genome sequencing of maternal plasma DNA: review of 1982 consecutive cases in a single center. Ultrasound Obstet Gynecol 2014;43:254–64.
[6] Porreco RP, Garite TJ, Maurel K, et al. Noninvasive prenatal screening for fetal triso-
mies 21, 18, 13 and the common sex chromosome aneuploidies from maternal blood using massively parallel genomic sequencing of DNA. Am J Obstet Gynecol Mar 19 2014. http://dx.doi.org/10.1016/j.ajog.2014.03.042 [Epub ahead of print].
[7] Benn P, Cuckle H, Pergament E. Non-invasive prenatal testing for aneuploidy: cur- rent status and future prospects. Ultrasound Obstet Gynecol 2013;42:15–33.
[8] Bianchi DW. Circulating fetal DNA: its origin and diagnostic potential—a review. Pla-
centa 2004;25:S93–S101 [Suppl. A].
[9] Alberry M, Maddocks D, Jones M, et al. Free fetal DNA in maternal plasma in anembryonic pregnancies: con?rmation that the origin is the trophoblast. Prenat Diagn 2007;27:415–8.
[10] Canick JA, Palomaki GE, Kloza EM, Lambert-Messerlian GM, Haddow JE. The impact of maternal plasma DNA fetal fraction on next generation sequencing tests for com- mon fetal aneuploidies. Prenat Diagn 2013;33:667–76.
[11] Taglauer ES, Wilkins-Haug L, Bianchi DW. Review: cell-free fetal DNA in the mater- nal circulation as an indication of placental health and disease. Placenta Feb 2014; 35:S64–8 [Suppl.].
[12] Wang E, Batey A, Struble C, et al. Gestational age and maternal weight effects on fetal cell-free DNA in maternal plasma. Prenat Diagn 2013;33:662–6.
[13] Ashoor G, Poon L, Syngelaki A, Mosimann B, Nicolaides KH. Fetal fraction in maternal plasma cell-free DNA at 11–13 weeks' gestation: effect of maternal and fetal factors. Fetal Diagn Ther 2012;31:237–43.
[14] Qu JZ, Leung TY, Jiang P, et al. Noninvasive prenatal determination of twin zygosity by maternal plasma DNA analysis. Clin Chem 2013;59:427–35.
[15] Leung TY, Qu JZ, Liao GJ, et al. Noninvasive twin zygosity assessment and aneuploidy detection by maternal plasma DNA sequencing. Prenat Diagn 2013;33:675–81.
[16] Martin A, Krishna I, Martina B, Samuel A. Can the quantity of cell-free fetal DNA pre- dict preeclampsia: a systematic review. Prenat Diagn 2014;34:685–91.
[17] Rava RP, Srinivasan A, Sehnert AJ, Bianchi DW. Circulating fetal cell-free DNA frac- tions differ in autosomal aneuploidies and monosomy X. Clin Chem 2014;60: 243–50.
[18] Schlütter JM, Hatt L, Bach C, Kirkegaard I, K?lvraa S, Uldbjerg N. The cell-free fetal DNA fraction in maternal blood decreases after physical activity. Prenat Diagn 2014;34:341–4.
[19] Liang D, Lv W, Wang H, et al. Non-invasive prenatal testing of fetal whole chromo- some aneuploidy by massively parallel sequencing. Prenat Diagn 2013;33:409–15.
[20] Hudecova I, Sahota D, Heung MM, et al. Maternal plasma fetal DNA fractions in preg- nancies with low and high risks for fetal chromosomal aneuploidies. PLoS One 2014; 9:e88484.
[21] Liang D, Peng Y, Lv W, et al. Copy number variation sequencing for comprehensive diagnosis of chromosome disease syndromes. J Mol Diagn 2014;16:519–26.
[22] Shaw SW, Hsiao CH, Chen CY, et al. Noninvasive prenatal testing for whole fetal chromosomal aneuploidies: a multicenter prospective cohort trial in Taiwan. Fetal Diagn Ther 2014;35:13–7.
[23] Blickstein I, Perlman S. Single fetal death in twin gestations. J Perinat Med 2013;41: 65–9.
[24] Benn P, Cuckle H. Theoretical performance of non-invasive prenatal testing for chro- mosome imbalances using counting of cell-free DNA fragments in maternal plasma. Prenat Diagn 2014;34:778–83.
[25] Chan KC, Zhang J, Hui AB, et al. Size distributions of maternal and fetal DNA in ma- ternal plasma. Clin Chem 2004;50:88–92.
[26] Yu SC, Chan KC, Zheng YW, et al. Size-based molecular diagnostics using plasma DNA for noninvasive prenatal testing. Proc Natl Acad Sci U S A 2014;111:8583–8.
 妇产科在线APP下载
妇产科在线APP下载













